探索癌症奥秘:3C 技术引领精准医学新征程
癌症,如同隐藏在生命深处的阴霾,始终是人类健康面临的巨大挑战。长期以来,科学家们不懈探索,试图揭开其神秘面纱,从传统病理学到现代分子生物学,每一次技术的突破都让我们离真相更近一步。而近年来, 3C 技术的兴起,尤其是 Hi-C 技术的迅猛发展,为癌症研究带来了前所未有的曙光。它宛如一把神奇的钥匙,开启了三维染色质组织这一神秘领域的大门,使我们得以窥探基因调控的深层奥秘,以及其在癌症发生发展过程中的关键作用。今天,就让我们一同走进这一前沿技术的世界,探寻它如何为癌症研究与治疗带来革命性的突破,引领精准医学迈向新的征程。

在细胞核内,三维(3D)染色质组织呈现出动态且复杂的特性,这一特性对基因调控、DNA 复制及细胞发育等多种细胞过程有着重要影响。其中,染色质环、拓扑相关结构域(TADs)以及像 CCCTC 结合因子(CTCF)和黏连蛋白等结构蛋白在塑造染色质结构中发挥着关键作用。3C 技术及其后续发展的 Hi-C 技术,为剖析 3D 染色质全景提供了可能,极大地推动了我们对基因组结构与功能的理解。
(一)原理概述
Hi-C 技术的核心在于能够捕捉并量化细胞核内基因组区域间的空间邻近性,这使得对关键染色质结构的全面研究成为可能。在哺乳动物细胞中,DNA 被组织成核小体,进而组装成更为复杂的染色质结构,这些结构在细胞周期、复制、发育和基因活性调控中发挥着重要作用。染色质结构内部存在着多种相互作用,如增强子 - 启动子相互作用可促进基因表达,而与核纤层相关结构域的相互作用则抑制基因表达。染色质结构的组织依赖于 CTCF 和黏连蛋白等关键结构成分。
 图1
图1
2.连接与片段分离: 通过连接酶处理,使空间上邻近的 DNA 片段连接起来。随后将连接后的 DNA 序列分离,用于构建测序文库。
3.测序与数据处理: 构建好的 Hi-C 文库采用高通量测序平台进行测序。原始测序读段需经过质量控制,去除低质量读段和接头序列,然后使用专门的比对算法将读段与参考基因组进行比对,确定其在基因组上的位置。在此过程中,Hi-C 片段可能会产生嵌合读段,未比对上的读段会进一步处理后整合到比对数据中。
4.数据分析: 对已比对的数据进行多方面分析,包括识别在原始样本中空间邻近的基因组位点对,计算位点对之间的接触频率并存储于交互矩阵中,还可进行变异检测、单倍型定相和 ecDNA 检测等操作。接触频率会进行归一化处理,以校正文库制备和测序过程中引入的偏差,最后将归一化数据可视化为接触图谱,利用生物信息学工具和算法分析接触图谱,识别染色质环、TADs、染色质区室、结构变异(SVs)和其他高阶染色质结构。
(一)Hi-C 数据分辨率提升技术
尽管目前多数 Hi-C 数据分辨率在 25kb - 1Mb 之间,但近年来多种计算技术致力于提高分辨率。例如,HiCPlus 运用超分辨率卷积神经网络算法,仅用 1/16 的原始测序读段就能生成与原始矩阵极为相似的交互矩阵;HiCNN 借助 54 层的深度卷积神经网络有效增强低分辨率 Hi-C 数据;Li 等开发的 SRHiC 基于深度学习,可从低分辨率矩阵推断高分辨率交互矩阵;HiDENSEC 作为分析异质性实体瘤的新计算框架,能预测拷贝数变异、大规模染色体重排和癌细胞比例。
(二)染色质构象捕获技术特异性改进
为提高染色质构象捕获技术的特异性,一系列方法应运而生。ChIP - loop 结合了 ChIP 和 3C 技术,用于鉴定蛋白质介导的染色质相互作用;ChIA - PET 采用富集策略,能在全基因组范围内鉴定特定蛋白质介导的染色质关联;PLAC - seq 和 HiChIP 通过应用原位 Hi-C 提高了 DNA 接触捕获效率和准确性,HiChIP 还采用 Tn5 转座酶介导的测序文库构建方法;ChIA - drop 则可通过液滴和条形码连接测序分离单个染色质复合物,无需连接步骤。
(三)单细胞技术在染色质结构分析中的发展
单细胞 Hi-C(scHi-C)技术不断发展,如对小鼠 CD4 + T 细胞的 scHi-C 分析揭示了细胞周期与染色质相互作用变化的相关性;单细胞核 Hi-C(snHi-C)技术在小鼠卵母细胞到受精卵过渡过程中成功重建了染色质结构;Dip - C 技术应用于单个人类二倍体细胞,通过转座子全基因组扩增获得单倍型数据。此外,单细胞多组学 Hi-C 技术也取得进展,如 scMethyl - HiC 和 sn - m3C - seq 可同时进行 Hi-C 和亚硫酸盐测序,HiRES、scCARE - seq 和 LiMCA 等技术能够同时检测染色质结构和基因表达,这些技术有助于在复杂组织或异质性癌症基因组中表征细胞类型特异性的染色质组织和表观基因组。
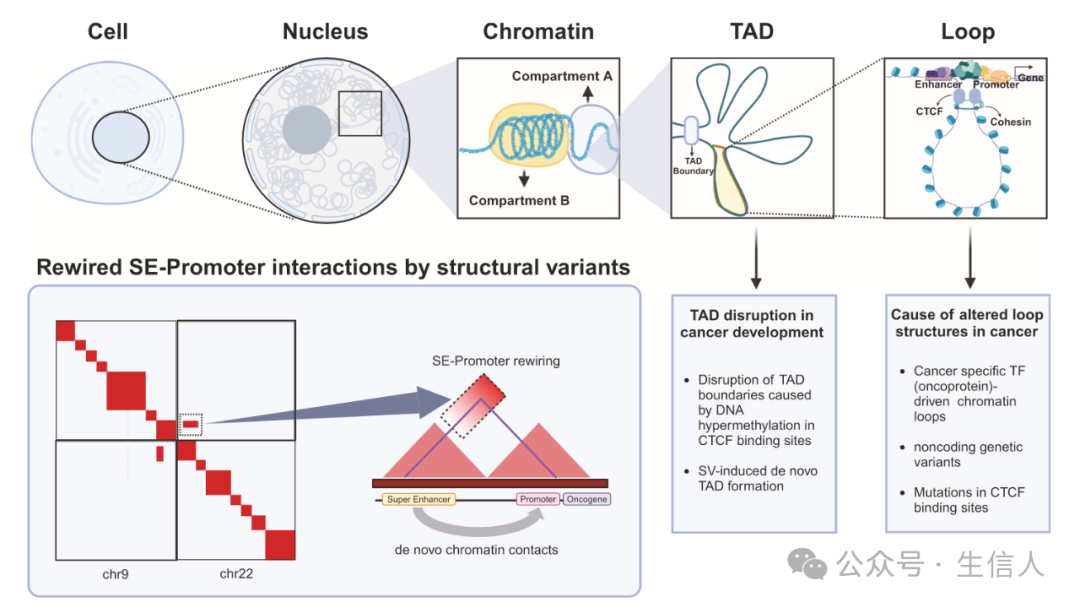 图2
图2
(一)染色质环
1.染色质环的定义与检测方法
染色质环在 Hi-C 数据中表现为相互作用增强的集中区域,对基因调控至关重要。传统的 Hi-C 等方法可检测长距离染色质环,而 HiChIP、TrAC looping 和 Capture - HiC 等新技术则专注于短距离环的高分辨率测序,提供更精确的染色质相互作用信息,包括增强子 - 启动子相互作用。Micro - C 和 Microcapture - C 等方法通过引入微球菌核酸酶提高分辨率,可用于研究人类癌症中的短距离染色质相互作用。
2.染色质环与癌症的关系
高分辨率染色质环研究为癌症研究带来新见解。在儿童恶性肿瘤如横纹肌肉瘤和尤因肉瘤中,转录因子(TF)结合在染色质环内发挥重要作用,融合阳性的横纹肌肉瘤亚型具有独特的由 PAX3 - FOXO1 癌蛋白驱动的染色质环,可作为潜在治疗靶点;融合阴性亚型的染色质环模式受临床药物影响。此外,TF 基序影响人类癌症中的调控染色质环,非编码遗传变异可改变染色质相互作用,FOXA1 和 GATA3 等先驱因子与乳腺癌和膀胱癌中的染色质环改变相关,CTCF 结合位点突变也会影响染色质环结构。
(二)结构变异
1.Hi-C 在检测结构变异中的作用
Hi-C 技术结合全基因组测序在检测人类基因组特别是癌症中的 SVs 方面表现出强大能力,可高效检测癌症基因组中的 SVs,如缺失、倒位和易位等,揭示其对基因调控相关的增强子和绝缘子的影响,以及在癌症中拷贝数变异(CNVs)和基因融合事件的拓扑背景,为理解肿瘤发生机制提供重要线索。
2.结构变异与癌症发生发展的关系
在白血病研究中,SVs 可改变 BCL11B 基因位点与其增强子的邻近性,影响基因表达。Hi-C 有助于理解 SVs 模式,如染色体碎裂,在其他方法难以有效捕获时发挥重要作用。SVs 诱导的增强子转位可调控多种癌症中的致癌驱动基因表达,SVs 与染色质相互作用的关联在癌症中日益明显,特定 SV 模式与癌症类型相关,其整合到转录调控元件和结构域中对癌症发展具有重要意义。
(三)TADs
1.TADs 的定义与功能
TAD 是基因组中自我相互作用增强的区域,由 CTCF 等主要调控因子界定边界,在胚胎发育过程中建立,对维持基因组正常组织和功能至关重要,参与细胞周期调控、DNA 复制等多种细胞过程,其失调与发育畸形和肿瘤发生相关。
2.TADs 与癌症的关联
癌症研究中,SVs 可破坏 TAD 边界,导致新的 TAD(neo - TAD)形成,激活癌基因或灭活肿瘤抑制基因,促进肿瘤发生。多种癌症如白血病、胃癌、乳腺癌和多发性骨髓瘤中均发现 TAD 破坏现象。此外,TADs 内的染色质纳米域(CNDs)具有重要结构意义,对其在增强子 - 启动子相互作用中的研究有助于深入理解癌症发展机制。
(四)CTCF
1.CTCF 的功能特性
CTCF 是高度保守的蛋白质,在调节 3D 染色质结构和基因表达方面发挥核心作用,参与转录调控、绝缘、基因组印记、DNA 修复和可变剪接等多种过程,其结合位点受表观遗传修饰、突变和环境因素影响,在胚胎发育早期的 CTCF 介导的环化过程高度保守,对细胞分化、命运决定和发育不可或缺,还能响应环境刺激。
2.CTCF 与癌症的联系
CTCF 异常与肿瘤发生相关,在胶质瘤中,突变可导致 DNA 高甲基化、TAD 边界破坏和癌基因激活;在前列腺癌中,CTCF 与其他蛋白质相互作用调控基因表达;在乳腺癌中,CTCF 与 EGR1 共同控制癌细胞迁移;在黑色素瘤中,CTCF 结合与 UV 损伤和突变热点相关。此外,CTCF 在人类发育疾病中也起重要作用,其在细胞衰老和肿瘤发生中的作用也备受关注。
(五)黏连蛋白
1.黏连蛋白的功能
黏连蛋白是染色体复合物中的结构维持蛋白,在细胞周期中确保姐妹染色单体的正确黏连,对维持有丝分裂染色体的结构至关重要,同时也参与维持间期染色体的 3D 结构,通过与 CTCF 等蛋白协作,调节染色质结构和基因表达,将染色质分隔为不同空间域,促进长距离相互作用和基因共调控。
2.黏连蛋白与癌症的关系
在癌症中,黏连蛋白相关蛋白失调与肿瘤发生发展相关,多种癌症中发现黏连蛋白基因编码蛋白的突变,其铰链域突变在癌症中起关键作用,黏连蛋白相关蛋白如 PDS5B 和 miRNA - 223 也与癌症有关,可作为潜在治疗靶点。针对异常黏连蛋白复合物的治疗策略有望成为癌症治疗新方向,黏连蛋白复合物基因的异常表达可作为癌症预后指标,在血液恶性肿瘤中,黏连蛋白突变影响 DNA 损伤修复和染色质结构,为治疗提供机会。
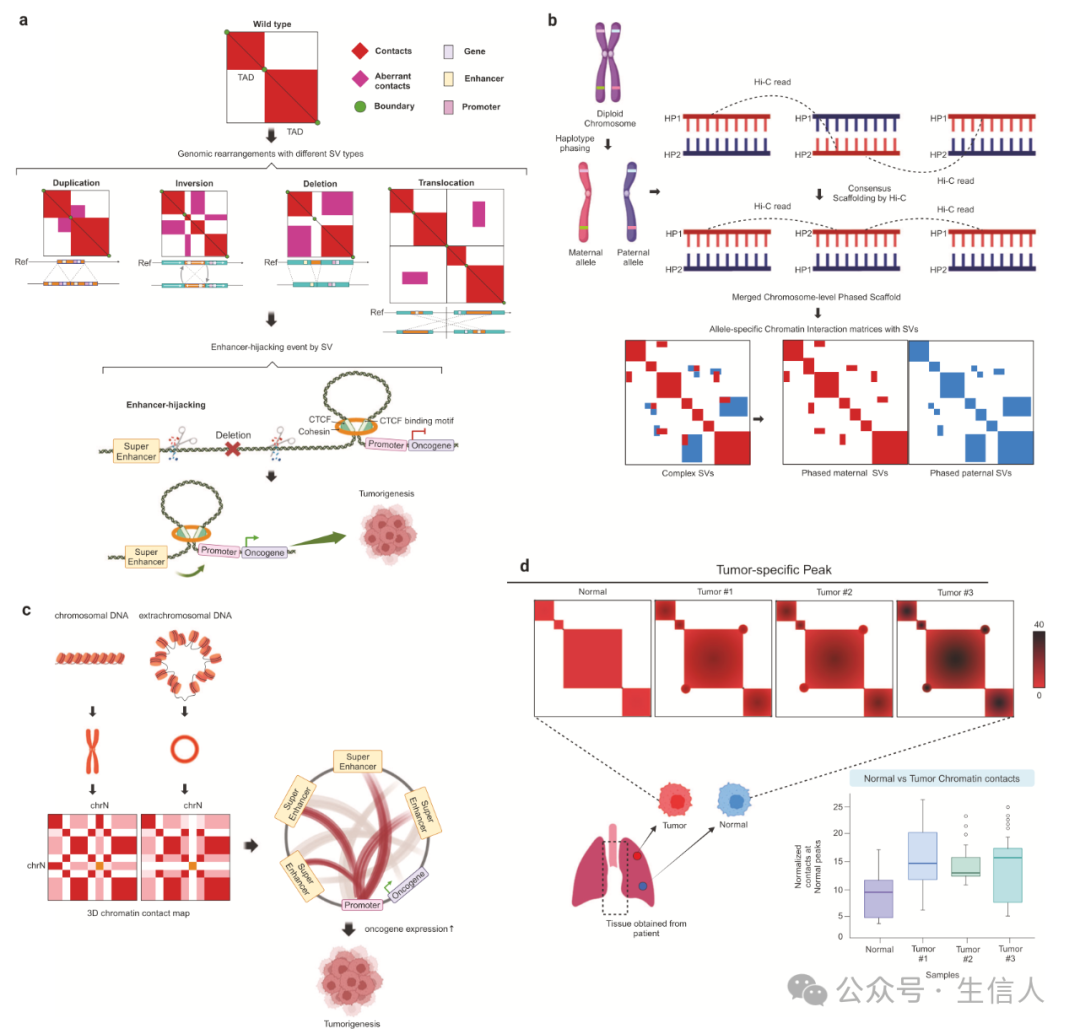 图3
图3 (六)驱动癌基因激活的结构变异检测
Hi-C 技术能够识别 TAD 内的癌症特异性改变,这些改变主要源于 SVs,可导致基因组 3D 组织破坏,即增强子劫持现象,激活癌基因,促进肿瘤细胞增殖和生长,在多种癌症如急性髓系白血病、神经母细胞瘤、髓母细胞瘤和结直肠癌中均有发现。超级增强子(SEs)也可通过增强子劫持促进癌基因表达,推动癌症发展。研究人员利用 Hi-C 数据结合 CRISPR - Cas9 基因组工程,能够预测癌基因激活情况,揭示癌基因调控网络的复杂性。
(七)单倍型水平的复杂基因组重排重建
Hi-C 数据在单倍型定相中发挥关键作用,单倍型定相可通过统计或实验方法进行,Hi-C 数据有助于组装初级重叠群并扩展为染色体水平的等位基因特异性重叠群,多种计算算法利用 Hi-C 数据实现这一目的。Hi-C 与长读长测序等技术结合,可提高定相变体的比例,有助于理解单倍型水平的复杂基因组重排,在癌症研究中,可识别等位基因特异性 SVs 和体细胞突变,深入了解体细胞克隆进化,为癌症诊断和治疗反应预测提供依据。
(八)ecDNA 检测
ecDNA 是存在于线性染色体外的环状 DNA 片段,大小从 kb 到 Mb 不等,在癌症中与肿瘤异质性、耐药性和不良预后相关,是癌症基因组多样性的重要因素。Hi-C 技术凭借 ecDNA 与染色体 DNA 的独特差异,如空间邻近性、形成嵌合 DNA 分子、独特 DNA 相互作用、SVs 和增强的信号等,能够有效检测和定位 ecDNA,为理解其在癌症中的作用提供关键信息,ecDNA 常作为移动增强子,扩增染色体转录,影响癌症发展。
(九)通过比较分析表征癌症中的 3D 染色质组织
染色质的 3D 结构在细胞过程中发挥重要作用,其破坏与癌症等疾病密切相关。研究发现,在乳腺癌中,CTCF 依赖的 3D 染色质组织变化与染色质可及性改变和转录失调相关,影响染色质区室、TADs 和染色质环,这些变化在癌症组织中部分保守。此外,药物耐药性或疗效也与 3D 染色质组织差异有关,理解药物对染色质组织的影响有助于揭示耐药机制,为癌症治疗提供新方向。
3C 技术通过揭示 3D 染色质相互作用的复杂性,包括染色质环、TADs 以及调控元件与癌基因的相互作用,彻底改变了我们对癌症生物学的理解,为癌症研究带来新的机遇。基于 3C 的技术如 HiChIP 和 Micro - C 为研究增强子 - 启动子相互作用和癌基因驱动的基因表达提供了高分辨率数据;3C 技术在检测癌症基因组中的 SVs 方面效果显著,有助于理解其对基因组调控区域的影响;Hi-C 技术在单倍型定相中的应用,结合长读长测序,能够精确识别等位基因特异性变异;Hi-C 技术对 ecDNA 的检测能力,有助于深入了解其在癌症发展中的作用;通过比较分析癌症中的 3D 染色质组织,揭示了其与表观遗传和基因表达变化的关联。尽管取得了进展,但仍面临挑战,如处理重复序列和改进结构变异映射方法等。未来,3C 技术将继续推动我们对癌症生物学的理解,为癌症的精准治疗提供有力支持。
链接:http://www.lewenyixue.com/2024/12/02/%E6%8E%A2%E7%B4%A2%E7%99%8C%E7%97%87%E5%A5%A5%E7%A7%98%EF%BC%9A3C%20%E6%8A%80%E6%9C%AF%E5%BC%95%E9%A2%86%E7%B2%BE%E5%87%86%E5%8C%BB%E5%AD%A6%E6%96%B0%E5%BE%81/