Nature | 单细胞追踪哺乳动物发育及癌前组织的演化过程
撰文: huacishu
IF=69.5
推荐度:
亮点:
1. 作者开发了一种多用途单细胞平台 NSC-seq ,用于同时捕获信使 RNA 和向导 RNA ( gRNA ),它利用 hgRNAs 的自突变 CRISPR 条形码通过累积突变模式进行谱系跟踪和时间记录;
2. 该平台为追踪哺乳动物发育及癌前组织的演化过程提供了强有力的工具。
范德堡大学医学中心 Ken S. Lau 教授课题组在国际知名期刊 Nature 在线发表题为 “ Temporal recording of mammalian development andprecancer ” 的论文。
细胞事件的时间顺序提供了对生物现象的基本见解。使用多用途的单细胞 CRISPR 平台,作者开发了一种分子钟方法,并结合细胞状态和谱系信息,来记录体内细胞事件和克隆性的时间。通过这种方法,作者发现了小鼠胚胎发育过程中组织特异性细胞扩增的精确时间,细胞类型与新的上皮祖细胞状态之间的非传统发育关系,以及它们独特的遗传史。对小鼠腺瘤的分析,结合人类癌前病变的多组学和单细胞分析,以及对 418 个人类息肉的克隆分析,表明 15-30% 的结肠癌前病变显示出多克隆起源。该研究将遗传变化与单细胞分析相结合,以探索哺乳动物系统发育和肿瘤发生的起源和时间。
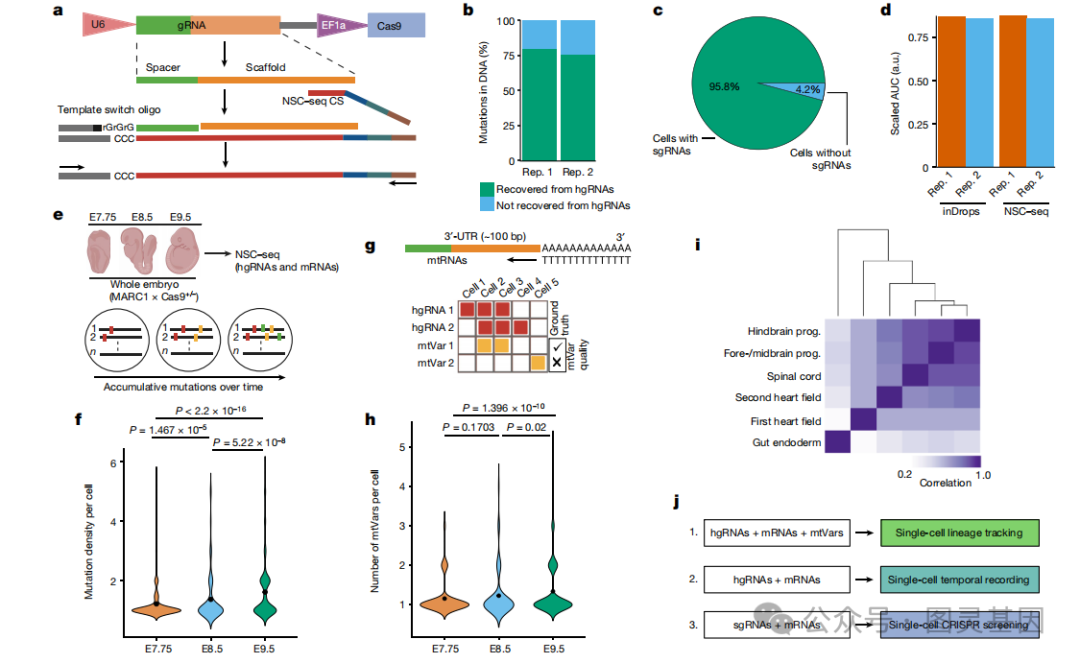
1 、时间记录平台
为了在单细胞分辨率下实现基于 CRISPR 的时间记录,作者开发了一种原生单向导 RNA 的捕获平台( NSC-seq ),该平台既不需要重新设计整个 gRNA 文库,也不需要间接读数(图 1a )。在具有 NSC-seq 的 hgRNA 中检测到近 80% 的 gDNA 突变(图 1b )。 NSC-seq 对单细胞分辨率的适应表明, 95% 的细胞中检测到 gRNA ,转录组质量与标准 inDrops 实验相似(图 1c , d )。
应用于三个小鼠胚胎时间点同时分析 hgRNAs 和信使 RNA 的 NSC-seq 显示出突变密度随时间增加(图 1e , f )。用体细胞线粒体变体( mtVars )增强 hgRNA 突变信息,使用自定义的 “ 种系 mtVars 库 ” 筛选出种系 mtVars ,然后从 mtVar 分布中定义了一个确定谱系的临界值。使用这条管道,作者发现 mtVars 在三个胚胎时间点上也持续增加(图 1g , h ),类似于 hgRNA 突变(图 1f )。使用 hgRNA 、 mtVars 或两者的单细胞分析能够准确地将淋巴细胞和髓系细胞识别为外周血单核细胞中的不同谱系,并区分胚胎组织类型(图 1i )。综上所述,研究结果证明了与单细胞转录组分析相结合的时间和谱系追踪的有效性(图 1j )。
图 1. 多用途单细胞捕获平台的优化
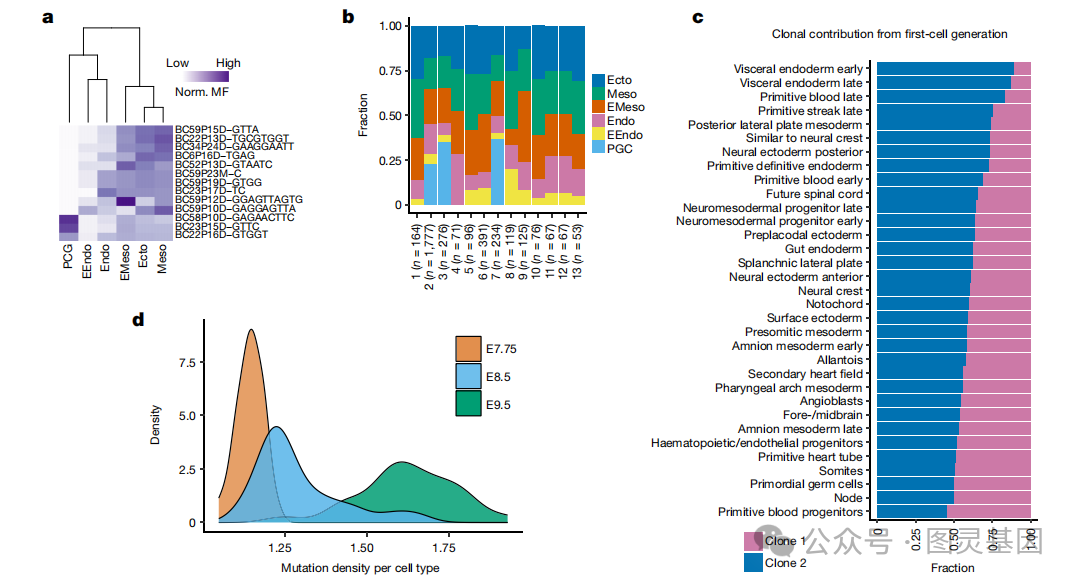
2 、谱系和细胞分裂追踪
通过分析早期胚胎突变( EEMs )回顾性地研究了发育的初始阶段,这些突变在最早的细胞分裂过程中表现出来,并由胚胎内的大部分细胞遗传。这些突变在细胞中的比例存在,称为镶嵌部分,是这些突变起源时细胞生成的指标。组织中共享的 EEM 的渐进限制使镶嵌组分能够用于模拟胚层和组织类型的早期分化(图 2a )。作者还评估了不同 EEM 对胚层(早期)或组织类型(晚期)的克隆贡献,并观察到不同早期克隆之间的贡献不均(图 2b )。作者发现第一代细胞克隆在不同组织类型中的分配不均(图 2c ),这表明早期胚胎祖细胞的特定谱系不是预先确定的,而是受潜在随机过程的影响。
图 2. 小鼠胚胎发生的谱系和时间记录
数据显示,在胚胎第 7.75 天( E8.5 )和 E9.5 阶段,组织特异性累积细胞分裂的分布越来越广,而在 E7.75 阶段观察到窄的单峰分布(图 2d ),这表明组织特异性细胞分裂和多样化始于 E7.75 期之后。总的来说,作者观察到原肠胚形成过程中造血祖细胞的高增殖,而心肌细胞和内皮细胞的增殖较低。与 E7.75 相比, E8.5 和 E9.5 的细胞类型间系统发育距离分布更广,支持 E7.75 后组织类型多样化的启动(图 2d )。这些数据强调了使用时间和谱系跟踪方法来获得早期哺乳动物发育和器官生成的新见解的稳健性。
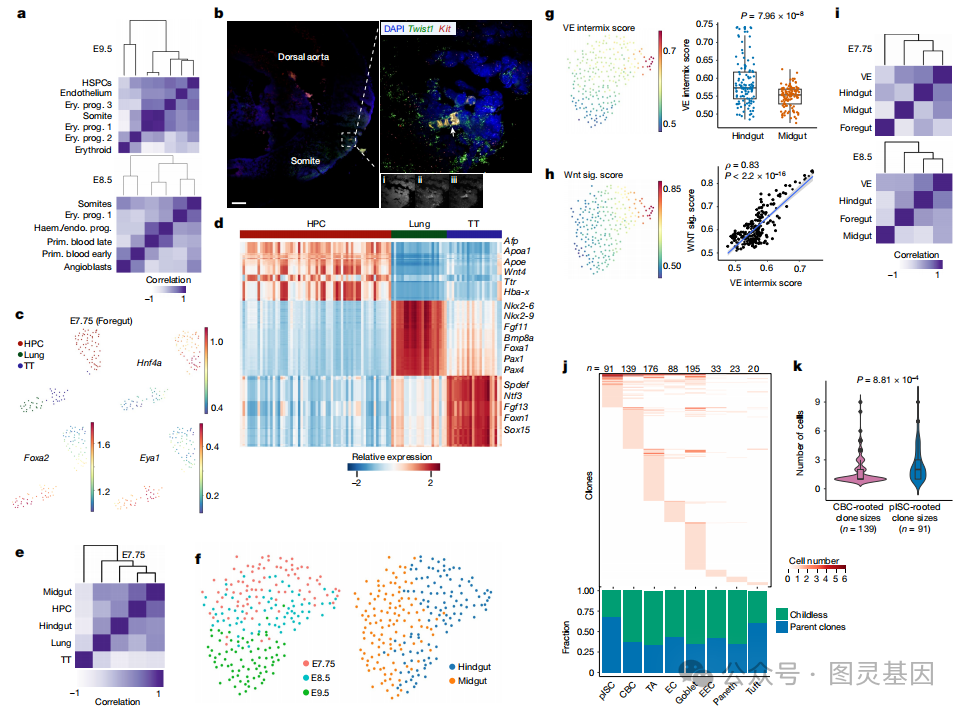
3 、非传统谱系多样化
作者重点介绍了在胚胎发育过程中发现的三个非传统谱系多样化的例子。 E8.5 和 E9.5 的谱系分析表明,红系祖细胞 1 ( EryPro1 )与体节细胞有共同的祖先(图 3a )。荧光原位杂交( FISH )显示, E9.5 胚胎体节区域有一簇 Kit + 红系细胞(图 3b ),支持体节衍生的红系祖细胞群。作者的数据显示,在哺乳动物发育的原肠胚形成晚期,有一个以前未被发现的体节来源的造血群体,与斑马鱼的造血群体相似。
图 3. 胚胎谱系多样化和肠道发育
接下来,作者试图在区域化和祖细胞鉴定的时间背景下了解肠道内胚层发育。对 E7.75 前肠群体的分析显示了三个不同的簇:肝胰腺( HPC )祖细胞( Hnf4a + )、肺祖细胞( Foxa2 + )和甲状腺 / 胸腺( TT )祖细胞的( Eya1 + )(图 3c )。基因表达、调节子活性和谱系分析表明, HPC 群体与肺和甲状腺 / 胸腺祖细胞相对不同(图 3d , e )。早在 E7.75 时期,中肠( Gata4 、 Pyyand-Hoxb1 )和后肠( Cdx2 、 Cdx4 和 Hoxc9 )祖细胞之间就存在不同的基因表达模式(图 3f )。这些结果表明,不同区域的祖细胞之间发育信号通路的早期差异使用,支持内胚层发育过程中的早期祖细胞规范模型。
内脏内胚层浸润特异性标记基因得出了内脏内胚层评分,确定了在发育中的后肠中显示出高内脏 / 定型内胚层混合的细胞(图 3g )。内脏内胚层混合评分与 Wnt 信号评分和 Wnt 反应基因( Lgr5 、 Axin2 和 Fzd10 )相关(图 3h )。使用突变条形码的谱系分析支持后肠和内脏内胚层之间的谱系关系,这可能是由于原肠胚形成过程中内脏内胚层衍生的细胞混合到后肠中造成的(图 3i )。这些数据证实内脏内胚层衍生细胞主要在后肠与定型内胚层混合,以及它们对肠道上皮发育的潜在贡献。
4 、肠道的持久祖细胞
人们普遍认为,以 Lgr5 标记的隐窝基底柱状细胞( CBC )是驱动成人肠上皮持续更新的稳态干细胞群,可能是肿瘤的起源细胞。然而,成人干 / 祖细胞的胚胎起源仍然难以捉摸。使用 NSC-seq 确定了一种与肠上皮细胞相关的独特细胞群,该细胞群从胚胎发育起源一直持续到成年,作者将这一群体称为持久性肠干细胞( pISC )。使用 hgRNA 突变的克隆贡献分析表明, pISC 群体具有更大的克隆大小,因此向肠上皮细胞贡献的后代比隐窝基底柱状细胞更多(图 3j , k )。这一发现支持了 pISC 群体在肠道发育过程中充当干 / 祖细胞样群体的前提。
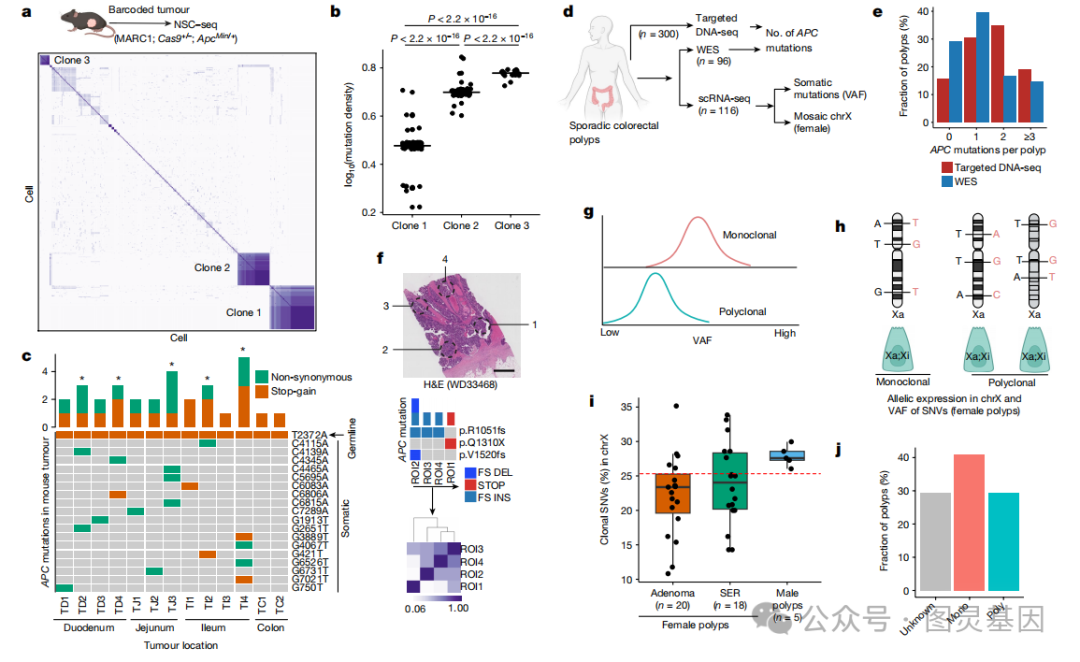
5 、结直肠癌前病变的克隆分析
肿瘤通常被认为是通过异常的发育基因程序形成的。癌症中一个悬而未决的问题是肿瘤是由单个干细胞还是由多个祖细胞产生,从而导致复杂的组织系统。因此,作者使用 NSC-seq ,类似于研究发育起源的方法,来研究肠道肿瘤发生的起源。
应用单细胞条形码追踪来描绘 Apc Min/+ 小鼠肠道肿瘤发生过程中的克隆性。为了描述克隆性,首先在淋巴细胞中使用了共享的条形码突变,证明与外周血淋巴细胞(大多为多克隆)相比,肿瘤浸润淋巴细胞已经克隆性扩增。类似的分析显示,肿瘤特异性细胞内有三个创始克隆(图 4a )。这三个克隆在许多特征上都是不同的,包括突变密度、克隆贡献、有偏分化和基因表达特征(图 4b )。作者发现, 13 只小鼠肠道肿瘤中有 5 只在 Apc 基因中有 3 个或更多独特的突变,这意味着存在多个创始克隆(图 4c )。
图 4. 结直肠癌前病变的克隆起源
尽管胚胎克隆混合只能用于遗传性疾病,但作者试图在人类散发性结肠癌前病变的两种最常见亚型中找到多克隆起始的证据。作者收集了新的 scRNA-seq 数据集,从范德堡大学医学中心( VUMC )的三个不同患者队列中总共收集了 116 个息肉数据集(腺瘤( AD ) 70 ;锯齿状息肉( SER ), 42 ;未知( UNK ), 4 )(图 4d )。使用田纳西州结直肠息肉研究数据,作者发现大约 20% 的息肉显示出三个或更多独特的 APC 突变,这意味着这些息肉中有多个创始克隆(图 4e )。
与这些结果类似, VUMC 息肉数据集的 WES 数据显示,大约 15% 的息肉发生了潜在的多克隆起始(图 4e )。在 VUMC 的 23 个结直肠癌( CRC )样本队列中使用已发表的多区域 WES 进行了 APC 突变分析,该队列显示只有一个样本表现出潜在的多克隆起始(图 4f )。
为了提供额外的克隆性证据,从结直肠息肉的单细胞转录组学数据中调用体细胞单核苷酸变异( SNVs )。然后使用体细胞 SNV 的变异等位基因频率( VAF )分布来评估克隆组成。如果息肉来源于单个创始克隆,其体细胞 SNV 的 VAF 分布将高于多克隆引发的息肉(图 4g )。
X 连锁转录物中的体细胞 SNV 可以用作发育标记,以追踪女性个体细胞的克隆起源(图 4h )。 X 连锁克隆 SNV 的比例随着多克隆程度的增加而降低。对相同规模的女性息肉的检查显示,大量息肉可能是多克隆性的(图 4i )。将 VAF 分布分析应用于所有息肉,发现大约 29% 是多克隆启动的(图 4j ),与基于 APC 突变的评估(图 4e )相当。因此,对多种数据类型的分析显示人类结肠直肠癌前病变的大量子集来自多个非癌祖先。
6 、结论
作者开发了天然单导向 RNA 捕获和测序( NSC-seq ),这是一种定制的多用途单细胞平台,用于同时捕获信使 RNA 和向导 RNA ( gRNA ),它利用 hgRNAs 的自突变 CRISPR 条形码通过累积突变模式进行谱系跟踪和时间记录。作者展示了该平台识别新的胚胎祖细胞群和细胞分化途径的能力,并为组织多样化的时间提供了新的见解。这些结果为各种应用的体内多模式记录奠定了基础。接着作者进一步利用这种追踪方法,将其与人类组织的基因组分析相结合,以阐明癌症的细胞起源。该多模式框架将人类的自然遗传变化与小鼠的诱导遗传变化配对,阐明了细胞起源和时间转换的复杂性,以及它们在早期肿瘤发生中的相关性。
教授介绍
Ken S.Lau 教授就职于范德堡大学医学中心,他的研究试图了解微环境因素,包括基质 / 免疫细胞和微生物群,如何在肠道炎症环境中影响上皮细胞行为。他的实验室利用高通量、单细胞和空间实验技术以及计算工具来研究类器官、小鼠模型和人类标本。试图从大型数据集获得生物学见解,以在炎症性肠病和癌症方面取得进展。
参考文献
Islam M, Yang Y, Simmons AJ, et al. Temporal recording of mammaliandevelopment and precancer. Nature. 2024;634(8036):1187-1195.doi:10.1038/s41586-024-07954-4
链接:http://www.lewenyixue.com/2024/11/20/Nature%20%7C%20%E5%8D%95%E7%BB%86%E8%83%9E%E8%BF%BD%E8%B8%AA%E5%93%BA%E4%B9%B3%E5%8A%A8%E7%89%A9%E5%8F%91%E8%82%B2/









赶快来坐沙发