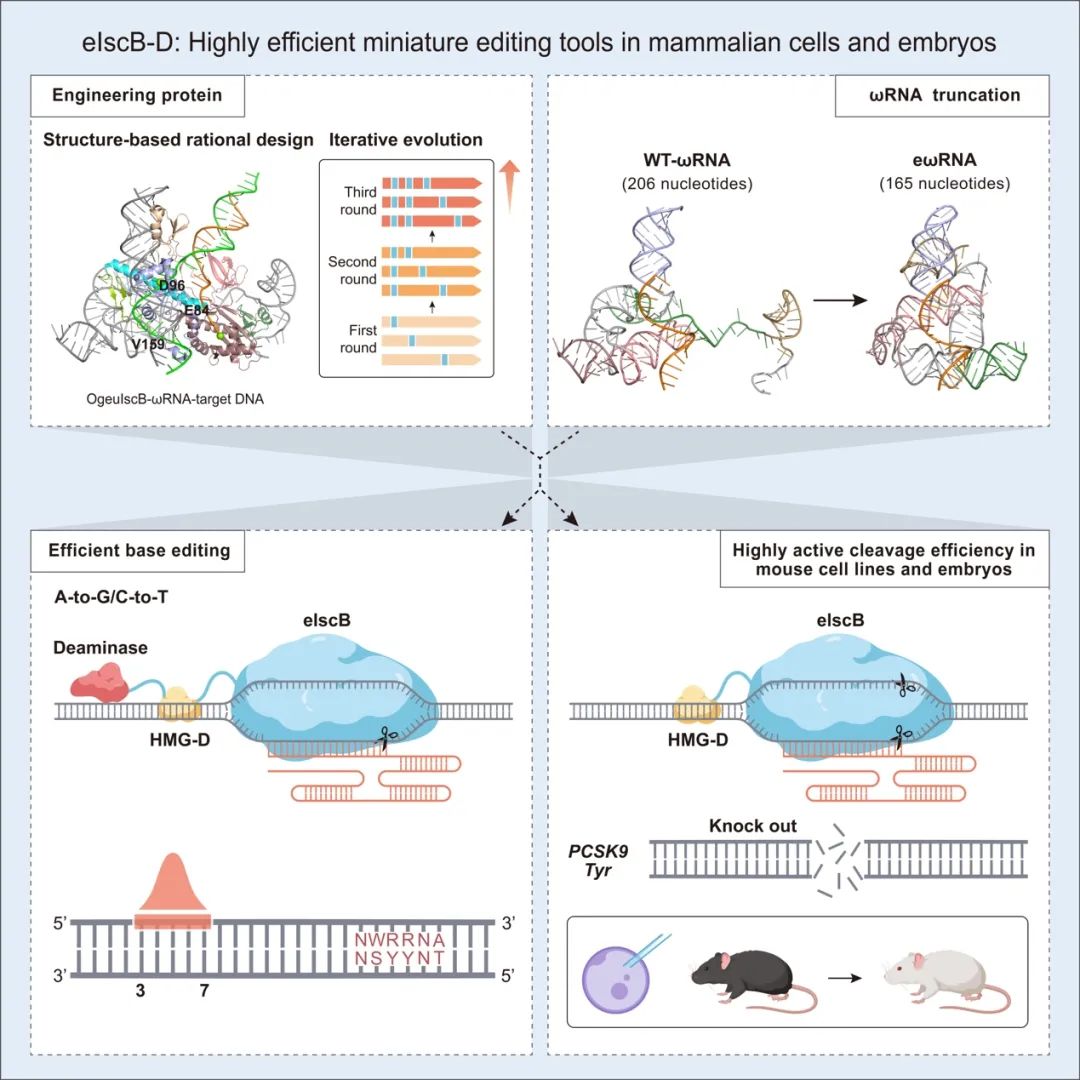
Mol Cell|实现小鼠体内高效编辑!李大力团队开发基于IscB的高活性迷你基因编辑工具

制版人:十一
参考文献
1. Wong, C. (2023). UK first to approve CRISPR treatment for diseases: what you need to know. Nature 623, 676–677.
2. Pausch, P., Al-Shayeb, B., Bisom-Rapp, E., Tsuchida, C.A., Li, Z., Cress, B.F., Knott, G.J., Jacobsen, S.E., Banfield, J.F., and Doudna, J.A. (2020). CRISPR-CasΦ from huge phages is a hypercompact genome editor. Science 369, 333-337.
3. Harrington, L.B., Burstein, D., Chen, J.S., Paez-Espino, D., Ma, E., Witte, I.P., Cofsky, J.C., Kyrpides, N.C., Banfield, J.F., and Doudna, J.A. (2018). Programmed DNA destruction by miniature CRISPR-Cas14 enzymes. Science 362, 839-842.
4. Chen, W., Ma, J., Wu, Z., Wang, Z., Zhang, H., Fu, W., Pan, D., Shi, J., and Ji, Q. (2023). Cas12n nucleases, early evolutionary intermediates of type V CRISPR, comprise a distinct family of miniature genome editors. Mol. Cell 83, 2768-2780.e2766.
5. Karvelis, T., Druteika, G., Bigelyte, G., Budre, K., Zedaveinyte, R., Silanskas, A., Kazlauskas, D., Venclovas, Č., and Siksnys, V. (2021). Transposon-associated TnpB is a programmable RNA-guided DNA endonuclease. Nature 599, 692-696.
6. Xiang, G., Li, Y., Sun, J., Huo, Y., Cao, S., Cao, Y., Guo, Y., Yang, L., Cai, Y., Zhang, Y.E., et al. (2023). Evolutionary mining and functional characterization of TnpB nucleases identify efficient miniature genome editors. Nat. Biotechnol. 42, 745-757.
7. Saito, M., Xu, P., Faure, G., Maguire, S., Kannan, S., Altae-Tran, H., Vo, S., Desimone, A., Macrae, R.K., and Zhang, F. (2023). Fanzor is a eukaryotic programmable RNA-guided endonuclease. Nature 620, 660-668.
8. Jiang, K., Lim, J., Sgrizzi, S., Trinh, M., Kayabolen, A., Yutin, N., Bao, W., Kato, K., Koonin, E.V., Gootenberg, J.S., et al. (2023). Programmable RNA-guided DNA endonucleases are widespread in eukaryotes and their viruses. Sci. Adv . 9, eadk0171.
9. Altae-Tran, H., Kannan, S., Demircioglu, F.E., Oshiro, R., Nety, S.P., McKay, L.J., Dlakić, M., Inskeep, W.P., Makarova, K.S., Macrae, R.K., et al. (2021). The widespread IS200/605 transposon family encodes perse programmable RNA-guided endonucleases. Science 374, 57-65.
10. Xu, X., Chemparathy, A., Zeng, L., Kempton, H.R., Shang, S., Nakamura, M., and Qi, L.S. (2021). Engineered miniature CRISPR-Cas system for mammalian genome regulation and editing. Mol. Cell 81, 4333-4345.e4334.
11. McGaw, C., Garrity, A.J., Munoz, G.Z., Haswell, J.R., Sengupta, S., Keston-Smith, E., Hunnewell, P., Ornstein, A., Bose, M., Wessells, Q., et al. (2022). Engineered Cas12i2 is a versatile high-efficiency platform for therapeutic genome editing. Nat. Commun . 13, 2833.
链接:http://www.lewenyixue.com/2024/08/03/Mol%20Cell%EF%BD%9C%E5%AE%9E%E7%8E%B0%E5%B0%8F%E9%BC%A0%E4%BD%93%E5%86%85%E9%AB%98%E6%95%88%E7%BC%96%E8%BE%91%EF%BC%81/