空间组学 + 肿瘤微环境:解锁精准抗癌新路径
在分析癌症患者的样本时,对肿瘤微环境(TME)的全面空间分析展示出巨大潜力,有望揭示疾病或耐药性的细胞和分子机制,并最终锁定潜在的药物靶点,用于疗法开发。
不过,在将空间转录组学方法引入您的项目时,应该从哪里入手呢?其他科学家使用这些方法的案例,尤其是在临床背景下研究人类肿瘤样本的案例,也许有助于您评估这些方法及其在癌症药物开发中的价值。
这篇文章探讨了空间转录组学在TME研究和癌症疗法开发中的新兴应用。它重点介绍了10x Genomics近期发布的一篇预印本,该论文采用Visium HD和Xenium原位分析以及Chromium单细胞基因表达Flex和单细胞免疫分析,研究人类FFPE结直肠癌样本。 这项研究体现了空间转录组学如何揭示TME中肿瘤细胞与免疫细胞之间复杂且与治疗相关的空间关系和相互作用,并为探究肿瘤边界的生物学特征提供了新的分析方法。
全面的TME空间图谱分析如何支持癌症药物发现与开发?
“探索癌症患者样本中各个细胞组分的存在和排列,为大多数实体瘤的诊断、分期以及制定治疗策略奠定了基础。”——Katie Shumake,《Stanford Medicine News》(2)
这种大胆的说法并非毫无道理。多项研究利用高分辨率的空间转录组学来绘制肿瘤细胞和免疫细胞在TME中的“存在和排列”,有力证明了空间生物学方法在转化肿瘤学中的新兴用途。
例如,最近的一项研究利用Xenium原位分析来表征乳腺癌亚型之间的肿瘤异质性,并鉴定T细胞和巨噬细胞与肿瘤细胞之间的相互作用,这些可作为免疫治疗积极应答的生物标志物(3)。同样,在一项使用新辅助治疗药物cabozantinib和PD-1抑制剂nivolumab治疗肝癌的1b期临床试验中,研究人员利用Visium空间基因表达发现癌旁免疫细胞区域中的B细胞活性增强,可作为积极治疗应答的生物标志物(4)。
还有一项研究利用Visium分析揭示了罕见的前列腺癌亚型与更常见的亚型在同一名患者肿瘤样本中共存的情况,为未来诊断检测和治疗方案的开发提供了指导(5)。最后,AACR 2024大会上的科研带头人证实了空间生物学在TME研究中的地位,他们讨论了如何利用空间转录组学数据和计算来更好地预测肿瘤进展和治疗应答。
高分辨率的空间转录组学技术在TME研究中有着广泛而创新的应用,有望促进癌症药物的发现与开发。下面列出了一些应用,尽管并不全面,但可以作为一个起点,帮助您考虑空间转录组学在哪些领域契合您的研究目标:
-
增强靶点的鉴定 :发现某种疾病状态下特有的新型细胞类型和相互作用,进而有望发现新的药物靶点。
-
加深对疾病机制的了解 :通过绘制空间背景下的细胞类型和相互作用,更深入了解疾病的进展过程,并确定病理学的关键驱动因素。
-
让药物筛选过程更具生理相关性 :对病变组织中细胞空间结构的了解有助于设计更精准的药物筛选体外模型(例如生成能够捕捉肿瘤空间要素的三维类器官模型,而不是简单的二维细胞‘片层’)。
-
生物标志物发现 :空间转录组学能够鉴定位置特异性的基因表达模式,这些模式可作为疾病进展或药物应答的新型生物标志物。
-
优化药物递送策略 :了解目标细胞的分布可为靶向药物递送系统的开发提供信息。
-
更好地预测药物疗效和副作用 :了解病变组织中复杂的细胞相互作用有助于预测潜在疗法的on-target和off-target效应。
-
降低耗损率 :对疾病机制和细胞相互作用的深入了解有助于您做出更明智的药物开发决策,减少后期代价高昂的失败。
利用高分辨率的空间技术探索结直肠肿瘤生物学的异质性
最近, 我们的科学家们发表了一篇bioRxiv预印本,该论文是将空间生物学工具应用于患者样本的TME转化研究的一个范例,也体现了使用空间转录组学可以获得的各种见解。 这项研究使用了一组人类FFPE结肠腺癌样本,包括肿瘤组织和癌旁正常组织(NAT)的FFPE组织块。为了深入研究这些样本,研究团队一开始通过Visium HD分析获得TME中细胞类型空间结构的“整体图像”,接着对肿瘤边界的组成进行了重点的结构特异性分析,最后还通过Xenium原位分析精细观察了巨噬细胞、T细胞和肿瘤细胞之间的相互作用。研究TME生物学时的这些分辨率水平让我们想到了一个类比,也许会有帮助:
探索肿瘤微环境就像探索一座城市。观看整个城市,尤其是从空中俯瞰,令人眼花缭乱,信息量巨大。您可以站在最高处观看城市中的各个部分和结构——摩天大楼、桥梁、公园和河流,了解其广泛的组织结构。数字地图则带给您完整的体验,既可以缩小后查看城市全貌,也可以放大后浏览街道和建筑,并且一直放大,直到看到这些结构上的砖块。
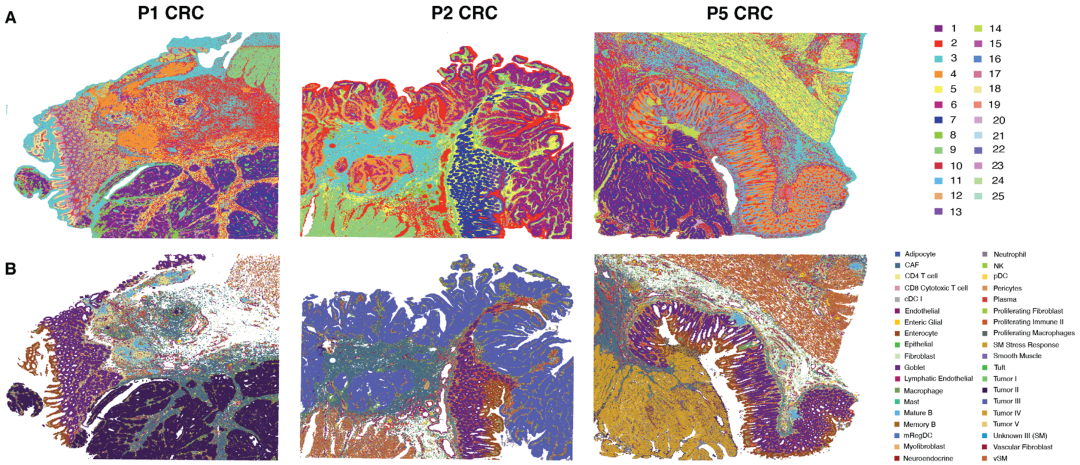
10x Genomics的科学家们主要利用Visium HD绘制出三位不同患者的结直肠肿瘤样本的同类图谱。Visium HD提供了组织切片上各个细胞中RNA的全转录组读数,通过空间条形码将转录本映射回其在组织中的精确位置,并将RNA读数叠加到组织的H&E图像上。这样可通过两种互补的分析方法,对肿瘤微环境中细胞组分不同的空间结构进行全面观察:1) 根据8 µm x 8 µm分析单位中的转录本组成来分配细胞类型,这种尺寸提供了单细胞尺度的空间分辨率(图1A);2) 使用相同样本的单细胞RNA测序参考数据集来预测(或解卷积)相同分析单位内的细胞类型(图1B)。
图1. 利用Visium HD绘制结直肠癌(CRC)样本的空间图谱可以揭示高分辨率且准确的转录本映射。A. 在8 µm分析单位上绘制三个CRC样本(P1 CRC、P2 CRC和P5 CRC)的空间图谱,根据无监督聚类进行着色。B. 在8 µm分析单位上绘制同样三个CRC样本的空间图谱,根据解卷积单细胞参考数据集后预测的细胞类型进行着色。图片来源:Oliveira MF等人论文中的图3A、B (1)。CC BY-ND 4.0.
为了确保他们的单细胞参考数据集能够严格代表空间分析所用的组织切片中的细胞类型,研究团队从同样三个FFPE组织块中采集了连续组织厚切片(与Visium HD所用的切片相邻),以及另外五个CRC和NAT组织块的厚切片。然后,他们利用Chromium单细胞基因表达Flex开展单细胞分析。 尽管Visium HD数据的无监督聚类本身就能提供有意义的结果,但使用单细胞参考数据集还有额外的好处,为不同患者肿瘤样本的细胞注释提供一致的策略,并支持罕见细胞类型的鉴定。 例如,解卷积后的Visium HD数据显示,三个患者样本中都存在独特的“主要”(或丰度最高)肿瘤细胞类型,这证实了肿瘤间异质性的存在,可能影响患者对治疗的应答。
Visium HD等空间转录组学方法非常有价值,因为能让研究人员更近距离地观察较大组织结构中特定结构的细胞组成,进而揭示细胞动态的重要信息,这些动态驱动了肿瘤的独特性质。 接下来,10x团队将空间分析的重点放在肿瘤边界的肿瘤细胞-免疫细胞相互作用上,因为这些细胞动态可以提供肿瘤侵袭潜力或免疫浸润能力的信息,因此可能对疾病进展和治疗应答产生重要影响。
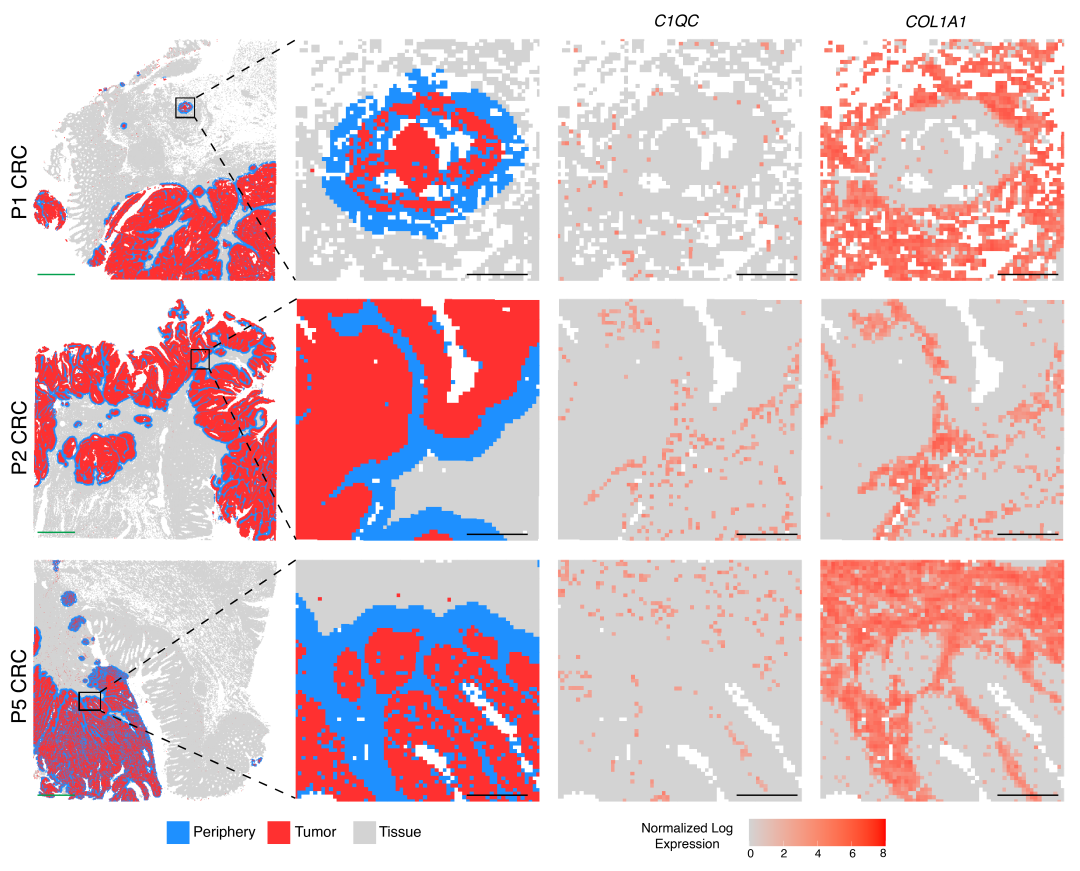
由于Visium HD数据可达到单细胞尺度的分辨率,研究团队 采用了基于距离的分析方法来解析肿瘤边界的细胞组成 ,他们首先选择了肿瘤细胞周边50 µm范围内的所有条形码标记的8 µm分析单位,然后定量了这些区域的细胞类型组成。 结果显示,癌症相关成纤维细胞是边界区域最主要的细胞类型,而巨噬细胞是最主要的免疫细胞类型。 研究团队根据成纤维细胞和巨噬细胞的已知标志物对其进行定位,证实了这一结果(图2)。
图2. 每张CRC切片中肿瘤周边的细胞组成。标注为肿瘤细胞的8 μm分析单位以红色显示,肿瘤周边50 µm范围的分析单位以蓝色显示。各行对应三个不同的样本。第一列显示6.5 x 6.5 mm的捕获区域,第二列显示放大后的视图,第三列显示C1QC(巨噬细胞)的对应表达,第四列显示COL1A1(成纤维细胞)的对应表达。图片来源:Oliveira MF等人论文中的图4A (1)。CC BY-ND 4.0.
巨噬细胞和肿瘤细胞在肿瘤边界的密切关联促使10x科学家对这一免疫细胞群体开展了更深入的研究。利用Visium HD提供的全转录组数据及其单细胞参考数据集,研究团队对肿瘤边界的8 μm分析单位进行了独立的无监督聚类。他们的分析表明,边界的一些细胞被分型为巨噬细胞。 他们进一步发现,这些聚类中存在两种不同的巨噬细胞亚群,其特征是表达SELENOP或SPP1基因。 他们还注意到,SELENOP+巨噬细胞表达的通路参与了NFK-β介导的TNF-α信号转导(已知在促进肿瘤方面发挥作用(6))。SPP1+巨噬细胞表达的基因则参与了凝血、胆固醇稳态和KRAS信号通路的上调(后者在癌症中往往过度活跃,导致细胞生长和存活失控(7))。
空间生态位分析表明,这些巨噬细胞亚群定位在整体基因表达谱不同的肿瘤区域,就像同一座城市中拥有不同特色的独特社区——不同的建筑、餐馆和文化。
在这个例子中,两个巨噬细胞“社区”似乎都存在促进肿瘤的基因表达特征。具体来说,SELENOP+ 巨噬细胞似乎与REG家族基因上调的肿瘤细胞共定位,REG家族基因通常与结直肠癌的转移、晚期阶段和预后不良有关。SPP1+ 巨噬细胞则与表达TGFBI的肿瘤细胞共定位,该基因也与预后较差有关(1)。这种对塑造肿瘤微环境特征的空间细胞关系的深入了解可为未来的患者分层提供方向,并揭示影响肿瘤进展或治疗应答的可能机制。
巨噬细胞只是塑造肿瘤微环境的免疫细胞类型之一。 接下来,研究团队试图探索T细胞在这些CRC样本中的定位和活性,特别是在肿瘤边界,因为招募肿瘤浸润淋巴细胞的能力对实体瘤的免疫治疗有影响。
首先,他们利用Visium HD数据鉴定了CRC样本中富含CD4+ 或CD8+ T细胞的区域,然后开展细胞核分割,并将转录本数据分配到与细胞核多边形对应的2 µm分析单位中的细胞核。结果显示,表达CD8A和CD4的T细胞零散分布在肿瘤边界,但未出现在癌旁正常组织中,表明这些T细胞可能在发挥抗肿瘤作用。
之后,为了验证Visium HD的结果,研究团队在CRC样本上使用了Xenium原位单细胞空间成像技术,这项技术以亚细胞分辨率分析一组精选基因。此次分析使用了预设计的Xenium人类结肠基因组合(322个基因)以及定制的附加基因组合(100个基因,靶向从Visium HD数据中鉴定出的免疫细胞)。他们的实验还采用了多模态细胞分割流程,通过边界染色和形态学对细胞进行分割。通过这种方法,研究团队确认REG1A+肿瘤细胞附近存在SELENOP+/STAB1+巨噬细胞,而TGFBI+肿瘤细胞附近存在SPP1+巨噬细胞。除了这些功能性细胞关系之外,他们还观察到表达MMP11的癌症相关成纤维细胞(MMP11表达的酶可分解细胞外基质,与预后较差相关)与TGFBI+肿瘤细胞定位相同,表明可能存在驱动肿瘤进展的细胞网络。
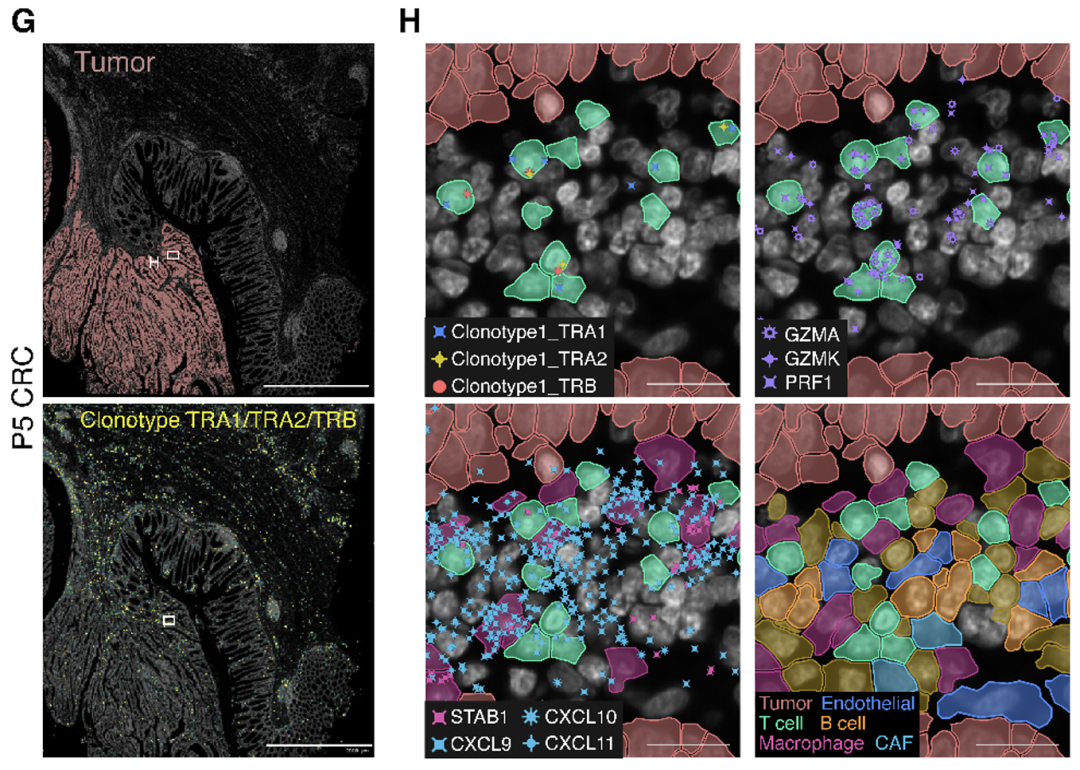
单细胞数据也发挥着强大的作用,能够完善和指引Xenium等超高分辨率的空间转录组学分析的见解。为了更好地了解T细胞活性和抗原识别,研究团队利用Chromium单细胞免疫分析(最近更名为通用型5’基因表达)来研究从患者样本中解离的肿瘤细胞。 他们发现一名患者(P5 CRC)的T细胞群中出现了扩增的T细胞克隆型(占总数的11%),它能识别该患者肿瘤特异性的新表位,这意味着这种T细胞受体(TCR)是全新的,并非来自之前的感染。
将这一发现与Xenium的功能相结合,研究团队在100个基因的定制组合中加入了靶向该克隆型TCR序列的探针,这样他们能够在整张P5 CRC肿瘤切片上定位该克隆型。 他们在肠道相关淋巴组织中发现了这些靠近肿瘤细胞的T细胞群,并通过基因表达的特征证实它们是CD8+细胞毒性T淋巴细胞。此外,这些T细胞定位在CXCL9/CXCL10/CXCL11趋化因子病灶以及STAB1+巨噬细胞*、B细胞和内皮细胞附近——这些细胞群很可能促进这些趋化因子的表达,进而将免疫细胞招募到肿瘤中。
* Xenium基因组合中不包含SELENOP,因此使用STAB1来定位该数据集中的巨噬细胞群体(详见图3H)。
图3. Xenium原位分析证实了巨噬细胞亚型和克隆扩增T细胞在肿瘤微环境中的存在和定位。G. 克隆型TRA1/TRA2/TRB在P5 CRC样本中的联合表达。H. 克隆扩增的CD8细胞毒性T细胞紧挨着肿瘤细胞并驻留在CXCL9/CXCL10/CXCL11病灶内。图片来源:Oliveira MF等人论文中的图7G、H (1)。CC BY-ND 4.0.
这些结果更完整地讲述了这名特殊患者肿瘤微环境中促肿瘤成分与抗肿瘤成分之间的战争。之前的数据表明,TME中存在的两个巨噬细胞亚群主要发挥促肿瘤作用,但研究团队此次发现了一个巨噬细胞亚群,它们是招募免疫细胞的趋化因子的主要来源,可能发挥了抗肿瘤作用。
利用单细胞和空间转录组学工具深入分析同一个肿瘤内这些相互竞争的生态位,这种能力代表我们在了解癌症生物学以及影响肿瘤进展和治疗应答的潜在细胞和分子机制方面取得了重要进展。 这些发现也为癌症药物开发提供了机会,例如以巨噬细胞为靶点,让它们转向抗肿瘤状态。
我们期盼,科学家们能够通过高分辨率且多重的空间转录组学工具提供的见解,更深入地了解癌症机制,克服异质性和耐药性,最终设计出更有效的疗法来治疗各种类型的癌症。
欢迎点击阅读原文,浏览我们的空间生物学概览页面,了解Visium和Xenium空间转录组学工具如何应用于前沿研究。
-
Oliveira MF, et al. Characterization of immune cell populations in the tumor microenvironment of colorectal cancer using high definition spatial profiling. bioRxiv (2024). https://doi.org/10.1101/2024.06.04.597233
-
Shumake K. The promise of spatial biology in understanding tumors. Stanford Medicine News (2024).
-
Wang N, et al. Spatial single-cell transcriptomic analysis in breast cancer reveals potential biomarkers for PD1 blockade therapy. Research Square (preprint) (2024). doi: 10.21203/rs.3.rs-4376986/v2
-
Zhang S, et al. Spatial transcriptomics analysis of neoadjuvant cabozantinib and nivolumab in advanced hepatocellular carcinoma identifies independent mechanisms of resistance and recurrence. bioRxiv (2023). doi: 10.1101/2023.01.10.523481
-
Watanabe R, et al. Spatial gene expression analysis reveals characteristic gene expression patterns of de novo neuroendocrine prostate cancer coexisting with androgen receptor pathway prostate cancer. Int J Mol Sci 24: 8955 (2023).
-
Wu Y and Zhou BP. TNF-α/NF-κB/Snail pathway in cancer cell migration and invasion. Br J Cancer 102: 639–44 (2010). doi: 10.1038/sj.bjc.6605530
-
Carvalho PD, et al. KRAS Oncogenic Signaling Extends beyond Cancer Cells to Orchestrate the Microenvironment. Cancer Res 78: 7–14 (2018). doi: 10.1158/0008-5472.CAN-17-2084
版权声明:本文为“乐问号”作者或机构在乐问医学上传并发布,仅代表该作者或机构观点,不代表乐问医学的观点或立场,不能作为个体诊疗依据,如有不适,请结合自身情况寻求医生的针对性治疗。
链接:http://www.lewenyixue.com/2025/03/21/%E7%A9%BA%E9%97%B4%E7%BB%84%E5%AD%A6%20%2B%20%E8%82%BF%E7%98%A4%E5%BE%AE%E7%8E%AF%E5%A2%83%EF%BC%9A%E8%A7%A3%E9%94%81%E7%B2%BE%E5%87%86%E6%8A%97%E7%99%8C%E6%96%B0/
THE END






赶快来坐沙发