前沿透视 | Cell:人工智能重新定义RNA病毒发现
“前沿透视”栏目聚焦新近最具影响力的医学科研成果,解读前沿突破,分享核心观点,旨在为广大科研工作者洞悉学术新知,激发创新思维提供新视角。
RNA病毒是一类以RNA作为遗传物质的病毒,它们在全球生态系统中具有广泛的宿主范围和显著的遗传多样性。这些病毒在动物、植物、真菌和水生环境jie中普遍存在,对生态系统健康和人类健康都有着重要影响。尽管RNA病毒的重要性已被广泛认识,但目前对它们的多样性了解仍不全面,许多RNA病毒尚未被发现。
传统的RNA病毒鉴定方法依赖于序列相似性比较和已知病毒数据库的完整性。这些方法在面对高度分化的RNA病毒时存在局限性,因为它们可能无法识别与已知病毒序列差异较大的新病毒。此外,现有工具可能错过一些高度分化的RNA依赖性RNA聚合酶(RdRPs),这是RNA病毒基因组的一个标志性成分。
人工智能(AI),尤其是深度学习算法,因其在准确性、性能、减少对特征工程的依赖以及自学习能力方面的优势,为RNA病毒的发现提供了新的可能性。AI能够处理更长的氨基酸序列,并有效捕捉序列间局部和长距离的关系,这使得它在识别高度分化的RNA病毒方面具有潜力。通过结合序列和结构信息,AI可以提高病毒鉴定的准确性和效率,从而加速新病毒的发现,拓展我们对RNA病毒多样性的认识。
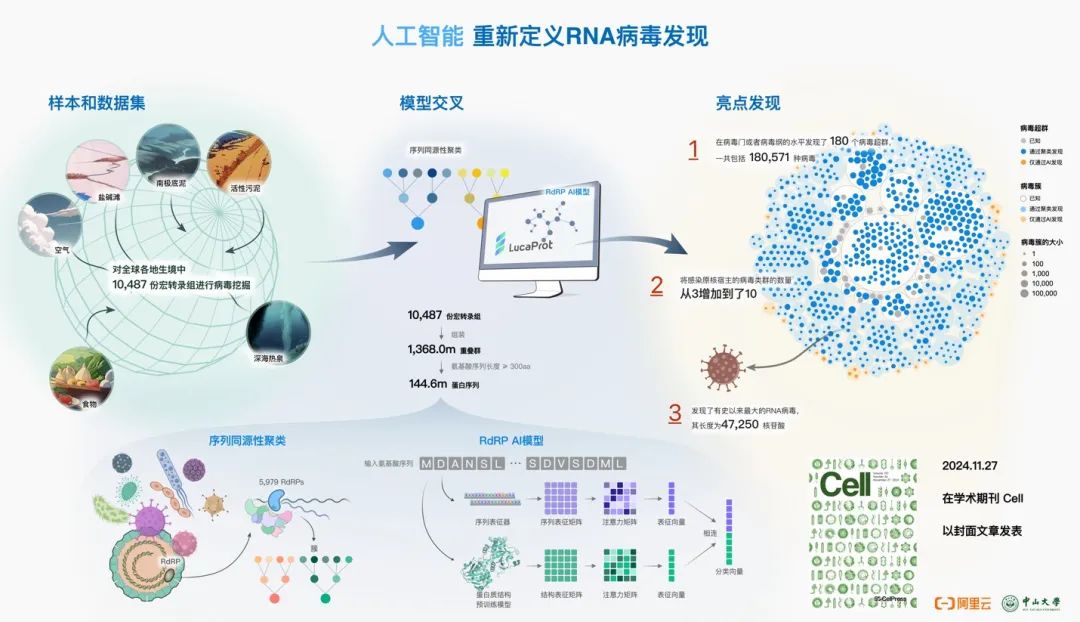
2024年11月27日,Cell期刊以封面文章发表了由中山大学医学院施莽教授团队和阿里云李兆融团队合作在Cell发表了题为“Using artificial intelligence to document the hidden RNA virosphere”的研究论文,报道了新开发的一种深度学习算法LucaProt。LucaProt集成了序列和预测结构信息,能够准确检测RNA依赖性RNA聚合酶(RdRP)序列。利用该方法,研究团队共确定了161979种潜在RNA病毒物种和180个RNA病毒超群,是已知病毒种类的近30倍,大幅提升了领域内对RNA病毒多样性和病毒演化历史的认知。
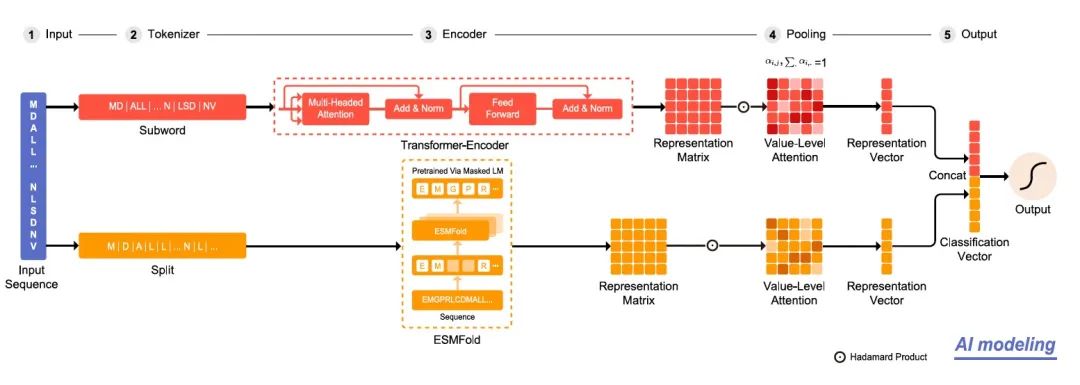
基于人工智能从序列+结构的角度对RNA病毒的RdRP进行鉴定
LucaProt模型从“序列+结构”两个维度入手,基于深度学习Transformer架构,从更高的维度去找可能的相似特征,极大提高了寻找的可能性和搜索空间。通过准备高质量的数据集,和采用双通路去提取序列和结构特征,LucaProt有效理解了RdRPs,并在各类数据集的比较里超过现有各种其他的模型方法。
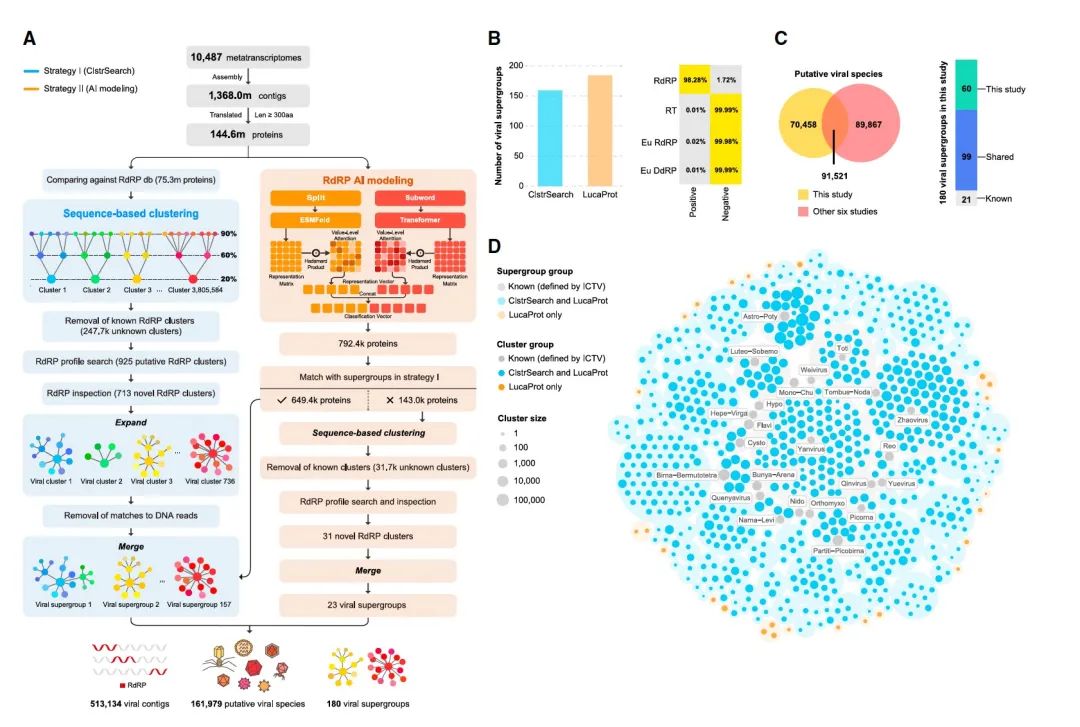
借助于高性能工具的研发,团队进一步对海量的宏样本进行地毯式的检索。研究团队收集了全球多种生态类型的10,487个环境样本的宏转录组测序数据,涵盖了包括水体、土壤、腐烂木材、根际以及极端环境(如热液喷口、盐湖、盐沼)等多种环境类型。同时,他们还自己在国内和南极洲采样,拿到了额外的50个珍贵的环境样本。
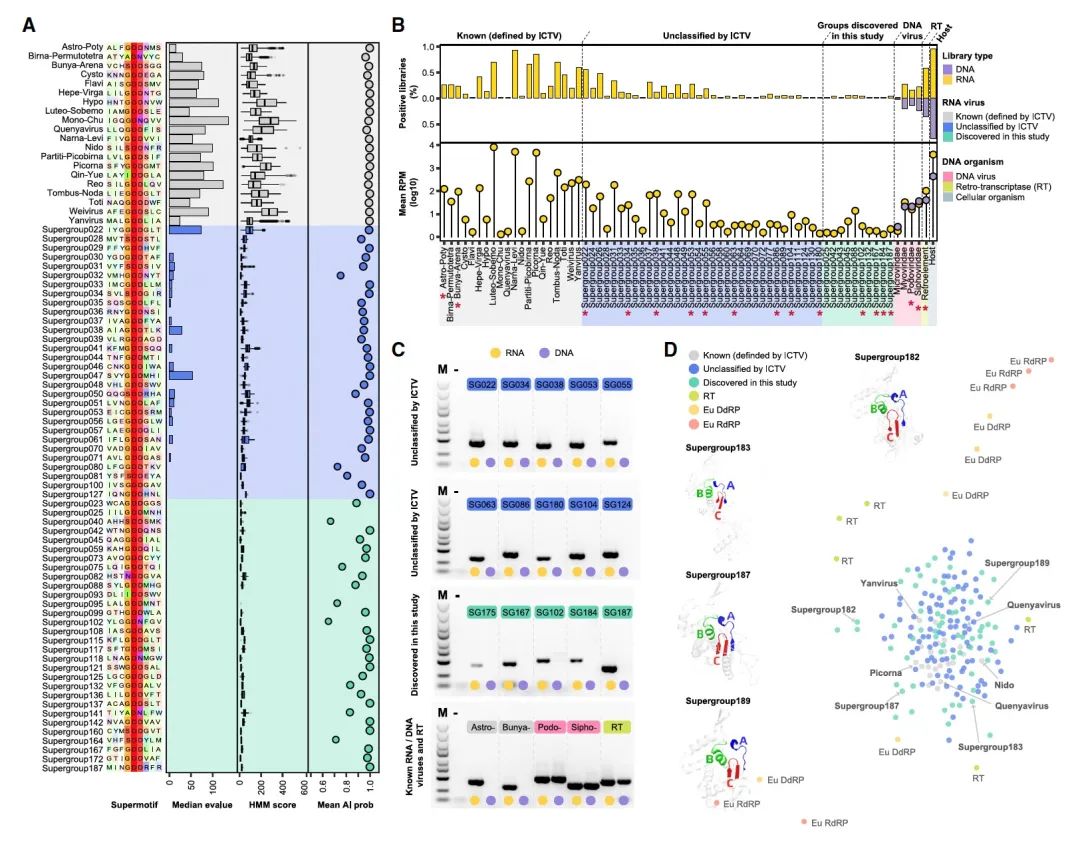
基于该数据集,研究人员使用LucaProt和ClstrSearch两种不同的策略揭示并交叉验证了潜在的病毒RdRPs,共发现了513134个RNA病毒组,代表了161979个假定的病毒物种以及180个RNA病毒超群。随后,对该研究和其他研究中具有统一定义的RdRP蛋白序列进行自动比较,发现LucaProt新鉴定的独特病毒共70458种。值得注意的是,研究还发现了60个以前未被识别和未被充分研究的病毒群体,并且其中23个病毒超群仅被LucaProt识别。
进一步,团队在模型预测产出的潜在病毒序列里,进行序列水平,结构水平,及实验的验证。例如对新发现的病毒超群进行了序列相似性分析,以确定它们是否为RNA病毒。这包括与细胞蛋白无相似性,并且具备关键的RdRP基序。通过三维比对,将新识别的病毒RdRP与已知的病毒RdRP、真核生物的RdRP(Eu RdRP)、真核生物DNA依赖的RNA聚合酶(Eu DdRP)和RT进行了结构相似性比较,显示出与已知RNA病毒蛋白相比显著更高的结构相似性。在实验中,对从南极等地采集的50多个样本同时进行DNA和RNA测序,验证他们发现的RNA病毒不是来自于DNA生物的污染。这确保了本研究工作产出的新的病毒类群的可信度。
作为一个典型的AI for Science的工作,该研究从一个明确的科学问题出发,通过模型创新更好的应用于RNA病毒发现这个研究目标。同时在严谨的进行各类工具对比后确认模型的领先性。对海量潜在样本进行系统性挖掘,最后进行各个角度的验证。将科学问题和人工智能手段进行了充分的融合。
Hou, X. et al. Using artificial intelligence to document the hidden RNA virosphere. Cell 187, 6929-6942.e16 (2024).
Shi, M. et al. Redefining the invertebrate RNA virosphere. Nature 540, 539–543 (2016).
Shi, M. et al. The evolutionary history of vertebrate RNA viruses. Nature 556, 197–202 (2018).
Koonin, E. V. The phylogeny of RNA-dependent RNA polymerases of positive-strand RNA viruses. Journal of General Virology 72, 2197–2206 (1991).
Koonin, E. V. et al. Global Organization and Proposed Megataxonomy of the Virus World. Microbiol Mol Biol Rev 84, e00061-19 (2020).
来源 | 科研办
阿里巴巴-浙江大学未来数字医疗 联合研究中心
编译 | 李兆融团队
版权声明:本文为“乐问号”作者或机构在乐问医学上传并发布,仅代表该作者或机构观点,不代表乐问医学的观点或立场,不能作为个体诊疗依据,如有不适,请结合自身情况寻求医生的针对性治疗。
链接:http://www.lewenyixue.com/2025/01/15/%E5%89%8D%E6%B2%BF%E9%80%8F%E8%A7%86%20%7C%20Cell%EF%BC%9A%E4%BA%BA%E5%B7%A5%E6%99%BA%E8%83%BD%E9%87%8D%E6%96%B0%E5%AE%9A%E4%B9%89/
THE END






赶快来坐沙发