前沿分享 | 人工智能驱动:从病理切片图像预测肺癌基因突变的重大突破
分享者导读
当下,人工智能发展迅猛,在医学领域中,深度学习与临床实践的结合也日益紧密。发表在 Lancet Oncology上的这篇文章展现了人工智能在肺癌基因突变预测方面的显著进展。该研究团队基于普通病理切片构建了DeepGEM模型,为精准医疗开拓了新方向。
引言
肺癌是全球致命癌症之一,每年夺走大量生命。 精准检测驱动基因突变对个性化治疗至关重要,然而传统基因检测技术存在样本要求高、成本高、耗时长等问题,在资源有限的地区难以推广。 在此背景下,该研究团队开发出DeepGEM这一人工智能模型,旨在通过病理切片图像预测基因突变,为精准医疗提供新途径。
研究背景
研究对象和数据特征
表1 参与者的基线特征
研究方法
研究结果与讨论
DeepGEM模型在多种场景下表现出色:
内部数据集: EGFR突变预测的AUC达0.95,准确率为91%,彰显其在熟悉数据中的精准预测能力。
表2 DeepGEM模型在内部预留测试集中预测基因突变的性能表现 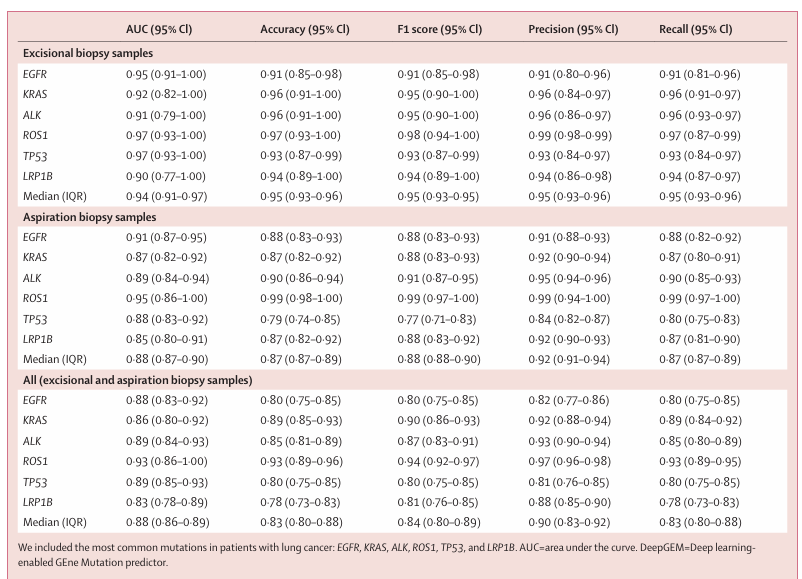
多中心数据集: 不同基因突变的AUC处于0.76-0.91之间,证明模型在不同环境下的可靠性和泛化能力。

图1 DeepGEM模型对多中心外部测试队列的泛化性能
淋巴结转移样本:EGFR突变的AUC为0.91,体现其处理复杂样本的能力。 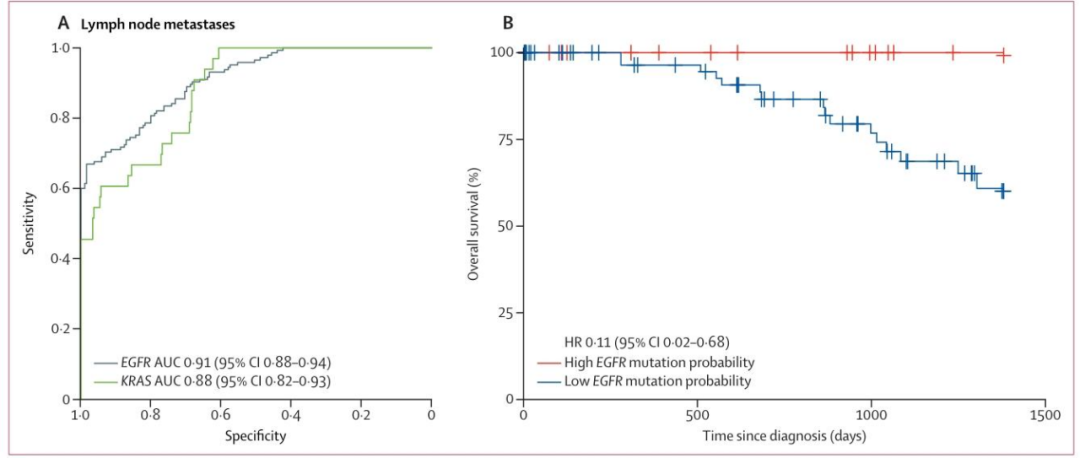
研究亮点
该研究开发了DeepGEM模型,可直接从常规组织学切片预测肺癌基因突变,为基因突变预测提供了全新途径。模型性能卓越,在内部数据集、多中心外部数据集以及TCGA数据集上,对多种常见基因突变(如 EGFR、KRAS等)的预测均展现出高AUC值和准确率,证明了其在不同环境和样本中的可靠性。该模型在临床应用方面潜力巨大,为肺癌患者提供了经济高效、及时准确的基因突变检测方法,有助于克服传统检测技术的局限,辅助医生制定个性化治疗方案,推动肺癌精准医疗的发展。
文献来源:
Zhao, Y., Xiong, S., Ren, Q., Wang, J., Li, M., Yang, L., Wu, D., Tang, K., Pan, X., Chen, F., Wang, W., Jin, S., Liu, X., Lin, G., Yao, W., Cai, L., Yang, Y., Liu, J., Wu, J., Fu, W., … Liang, W. (2024). Deep learning using histological images for gene mutation prediction in lung cancer: a multicentre retrospective study. The Lancet. Oncology, S1470-2045(24)00599-0. Advance online publication. https://doi.org/10.1016/S1470-2045(24)00599-0
原文链接: https://www.sciencedirect.com/science/article/pii/S1470204524005990?via%3Dihub
分享者简介
重庆大学智能肿瘤学教育部医药基础 研究创新中心2024级基础医学硕士
链接:http://www.lewenyixue.com/2025/01/02/%E5%89%8D%E6%B2%BF%E5%88%86%E4%BA%AB%20%7C%20%E4%BA%BA%E5%B7%A5%E6%99%BA%E8%83%BD%E9%A9%B1%E5%8A%A8%EF%BC%9A%E4%BB%8E%E7%97%85%E7%90%86%E5%88%87%E7%89%87%E5%9B%BE/








赶快来坐沙发