前沿分享 | SPRINTER算法:解锁肿瘤克隆增殖的进化密码

癌症的增殖是其关键特征之一,然而,对于同一肿瘤内不同克隆间的增殖是否存在差异,此前并不明确。来自英国伦敦大学学院癌症研究所的Simone Zaccaria等研究人员,在 Nature Genetics发表了题为“Characterizing the evolutionary dynamics of cancer proliferation in single-cell clones with SPRINTER”的文章,介绍了一种名为SPRINTER的算法,该算法通过对单细胞全基因组DNA测序数据的分析,能够准确识别处于S期和G2期的细胞,并将其分配到不同的肿瘤克隆中,从而为估计克隆特异性增殖率提供了有效手段。

增殖在癌症发展中起着关键作用,且与多种肿瘤的不良预后相关。以往主要通过病理或实验技术测量肿瘤样本中S期细胞比例来估计增殖,但肿瘤存在异质性,不同克隆具有不同进化历史和作用,且此前缺乏联合测量克隆特异性增殖率和相关进化动力学的方法。单细胞全基因组DNA测序技术虽可对肿瘤克隆进行基因组和进化分析,但在识别S期细胞及克隆分配方面仍面临挑战。
研究对象:
主要为非小细胞肺癌(NSCLC)患者的肿瘤样本,包括来自患者CRUKP9145的10个肿瘤样本(5个原发肿瘤样本和5个转移瘤样本),以及7例三阴性乳腺癌(TNBC)和15例高级别浆液性卵巢癌(HGSC)肿瘤样本。
1.对NSCLC样本进行单细胞全基因组DNA测序,共获得14,994个细胞的数据,其中原发肿瘤9,532个细胞,转移瘤5,462个细胞。
2.TNBC和HGSC数据集分别包含42,009个和19,905个细胞的数据。
1.开发SPRINTER算法: 利用scDNA-seq数据,通过计算复制特异性DNA测序信号、进行复制感知的基因组分割、高灵敏度推断S期细胞、推断不同肿瘤克隆、将S期细胞分配到相应克隆以及推断G2期细胞等6个步骤,实现对S 期 和G2期细胞的准确识别和克隆分配。算法利用已知的基因组区域复制时间信息,结合统计方法,克服了以往方法在S期细胞克隆分配和识别中的局限性。
2.生成基准数据集:采用基于EdU的FACS方法,对来自HCT116结直肠癌细胞系的8,844个二倍体和四倍体细胞进行分选,使其进入不同细胞周期阶段,然后使用DLP+进行测序,以评估算法性能。
3.生物信息学分析:将生成的数据集与人类参考基因组hg19比对,使用标准scDNA-seq管道处理,得到单细胞伪批量BAM文件。应用SPRINTER算法对基准数据集和NSCLC样本数据进行分析,同时应用其他相关方法进行比较。
4.系统发育和转移播种分析:利用SNVs和CNAs对NSCLC数据集中的克隆进行肿瘤系统发育重建,采用三步法整合并扩展现有方法,包括使用伪批量方法和概率模型推断每个克隆中的SNVs,使用HUNTRESS算法重建SNV进化,使用MEDICC2算法重建CNA进化,并基于此应用MACHINA算法推断转移迁移模式和识别播种克隆。
5.识别克隆特异性ART:基于SPRINTER的结果,通过比较克隆与正常细胞的复制时间分类,识别肿瘤克隆中的克隆特异性ART,并通过整合匹配的批量RNA测序数据进行基因集变异分析和差异基因表达分析,以支持ART分类。
6.分析ctDNA:利用先前研究中患者CRUKP9145的ctDNA数据,将跟踪的SNVs与SPRINTER识别的单细胞克隆进行匹配,计算每个克隆的ctDNA脱落指数,以评估克隆增殖与ctDNA脱落的关系。
7.计算克隆特异性基因组变异率:在TNBC和HGSC数据集中,根据先前研究中识别的变异,计算单个细胞中克隆特异性SNVs、SVs和CNAs的比率,并根据推断的S分数将克隆分为高增殖和低增殖两组,比较两组间变异率的差异。
8.分析高增殖克隆中的基因组改变:采用假设检验方法,识别TNBC和HGSC数据集中高增殖克隆中富集的已知癌基因扩增、肿瘤抑制基因缺失和驱动突变,并进行基因集富集分析。
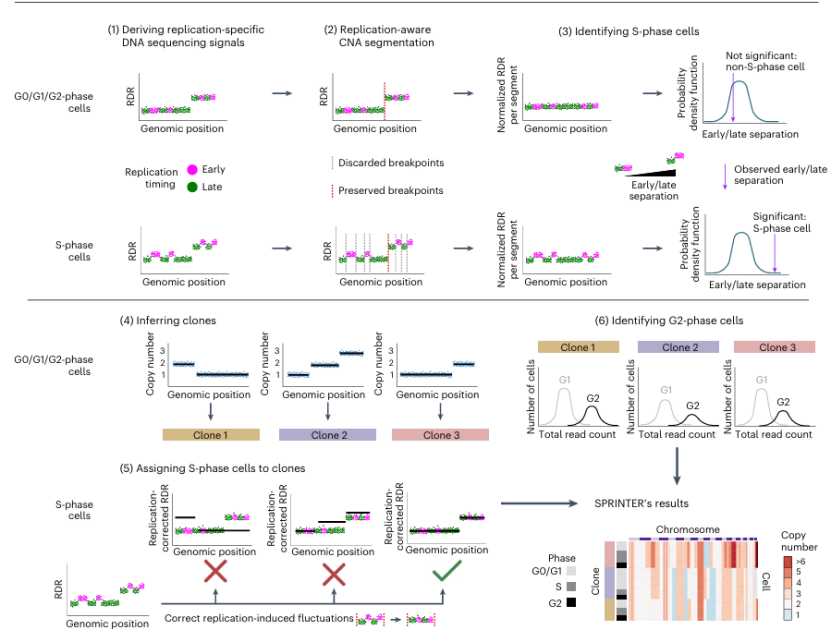
图1 SPRINTER算法
SPRINTER算法性能优异:
(1)在识别S期细胞方面,SPRINTER优于CCC和MAPD等先前方法,在中晚期S期识别上准确率提高10%-90%,且在不同倍性细胞混合的数据集中表现稳健,能准确识别G2期细胞;
(2)能准确区分克隆间的增殖率差异,所需复制细胞数量少,在克隆分配上准确性高,可准确推断S期细胞的CNAs。
肿瘤克隆增殖存在异质性:
(1)在NSCLC样本中,不同样本间及样本内克隆间增殖率存在广泛差异,且SPRINTER的克隆特异性估计能发现这些差异,而传统的整体估计方法(如Ki-67分析)则无法做到;
(2)利用Ki-67分析、DLP+核成像和临床成像等正交方法验证了SPRINTER结果的可靠性,如Ki-67染色与SPRINTER的增殖估计总体一致,SPRINTER的相位预测与核直径相关,其估计的平均S分数与肿瘤生长率相关。
增殖与转移相关:
(1)在NSCLC患者中,高增殖克隆具有更高的转移播种潜力,如第三个转移性分支包含最增殖的克隆,且是唯一包含额外播种克隆的分支,其播种遗传距离与S分数呈负相关;
(2)在TNBC和HGSC数据集中,高增殖克隆中细胞的单克隆特异性SNVs、SVs和CNAs比率显著高于低增殖克隆,且与细胞分裂相关的基因改变在高增殖克隆中富集,如CDK4和EGFR等癌基因扩增;
(3)高增殖克隆的ctDNA脱落指数更高,表明其与ctDNA脱落相关。
增殖与ART相关:
(1)在NSCLC中,最增殖和传播的转移性分支存在特定的ART事件(如KRAS的晚期到早期ART),且与基因表达变化相关,尽管未发现该分支特有的遗传驱动因素,但ART为癌症进展提供了一种非遗传进化机制。
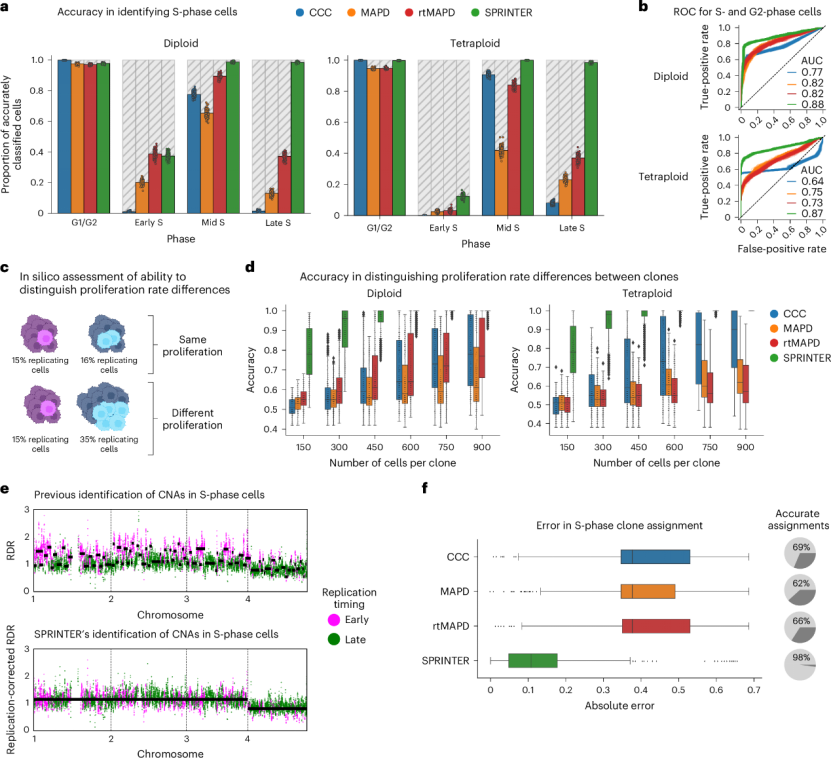
图2 SPRINTER改进了S期鉴定,并实现了S期细胞的准确克隆分配
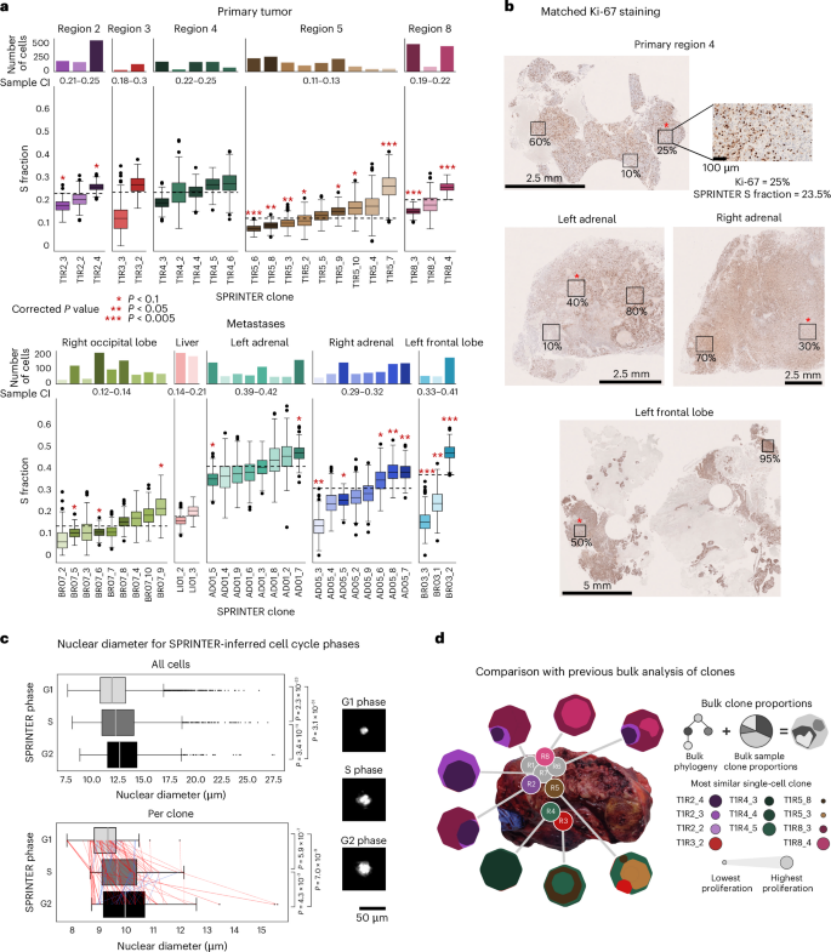
图3 SPRINTER鉴定NSCLC患者CRUKP9145的肿瘤克隆增殖异质性
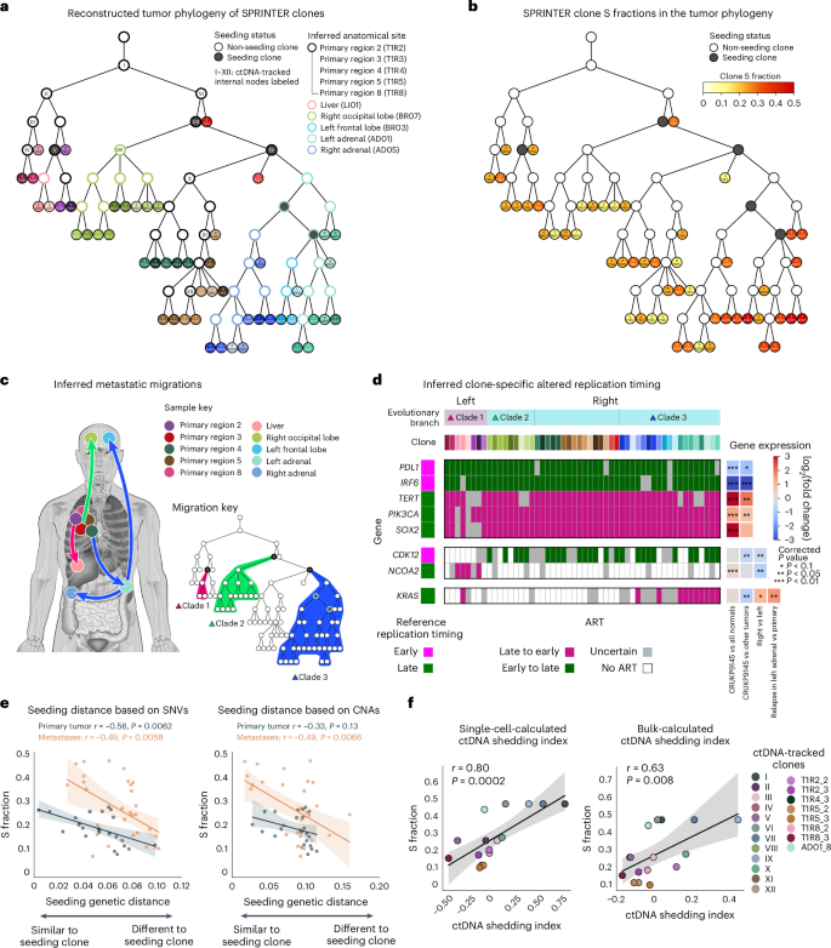
图4 SPRINTER揭示了克隆增殖和转移播种之间的联系,以及存在于不同转移分支中的克隆特异性ART
1. 开发了SPRINTER算法,能准确识别和分配肿瘤克隆中的S期和G2期细胞,评估克隆特异性增殖率。
2. 揭示了肿瘤内克隆间增殖率的异质性,高增殖克隆具有更高的转移播种潜力、特定基因改变和ctDNA脱落能力。
3. 发现增殖与ART相关,为研究癌症进展的非遗传机制提供了方法。
文献来源:
Lucas O, Ward S, Zaidi R, Bunkum A, Frankell AM, Moore DA, Hill MS, Liu WK, Marinelli D, Lim EL, Hessey S, Naceur-Lombardelli C, Rowan A, Purewal-Mann SK, Zhai H, Dietzen M, Ding B, Royle G, Aparicio S; TRACERx Consortium; PEACE Consortium; McGranahan N, Jamal-Hanjani M, Kanu N, Swanton C, Zaccaria S. Characterizing the evolutionary dynamics of cancer proliferation in single-cell clones with SPRINTER. Nat Genet. 2024 Nov 29. doi: 10.1038/s41588-024-01989-z. Epub ahead of print. PMID: 39614124.
文献链接:
https://www.nature.com/articles/s41588-024-01989-z
分享者简介 ✦
重庆大学智能肿瘤学教育部医药基础研究创新中心2024级生物与医药硕士
编辑|曾海蓉 李劲
版权声明:本文为“乐问号”作者或机构在乐问医学上传并发布,仅代表该作者或机构观点,不代表乐问医学的观点或立场,不能作为个体诊疗依据,如有不适,请结合自身情况寻求医生的针对性治疗。
链接:http://www.lewenyixue.com/2024/12/09/%E5%89%8D%E6%B2%BF%E5%88%86%E4%BA%AB%20%7C%20SPRINTER%E7%AE%97%E6%B3%95%EF%BC%9A%E8%A7%A3%E9%94%81/
THE END













赶快来坐沙发