Cell Discovery | 陈盛泉/陈洛南团队合作揭示单细胞预训练模型中潜在的安全风险:后门威胁
海量单细胞数据的积累给AI for Science研究,特别是单细胞预训练模型研究带来了新的契机,scBERT [1] 、GeneFormer [2] 、scGPT [3] 、scFoundation [4] 等模型相继被提出,在细胞类型注释、基因调控网络推断等多种重要任务上表现优异。然而,由于数据采集和模型训练中无意失误或刻意投毒等行为,单细胞预训练模型可能面临后门攻击的威胁,影响其在生物医学研究中的可靠性和准确性。
近日,南开大学数学科学学院 陈盛泉副教授团队 、中国科学院分子细胞科学卓越创新中心 陈洛南研究员 合作在 Cell Discovery 发表题为“Unveiling potential threats: backdoor attacks in single-cell pretrained models”的通讯文章(Correspondence), 首次提出了单细胞预训练模型后门攻击策略scBackdoor,在scGPT、GeneFormer、scBERT等多个单细胞预训练模型上进行了实验,攻击成功率均接近100%,表明现有的主流单细胞预训练模型普遍存在后门攻击风险,给单细胞研究特别是采用公开数据的AI预训练模型研究带来了严重的潜在威胁。
后门攻击是一种通过恶意篡改训练数据或模型,使模型在特定触发条件下输出攻击者指定结果的攻击方式。这种攻击 能够 在不影响 模型 正常功能的情况下隐藏恶意行为,严重威胁模型的安全性和研究的可信度。 该 通讯文章提出了 后门攻击策略 scBackdoor,以 评估现有单 细胞预训练模型在面对后门攻击时的脆弱性 。 以细胞类型注释任务为例,后门 攻击的目标 是 使得中毒模型 (被植入后门的模型) 在输入正常 的测试样本 时表现良好, 具有和正常模型相同的 细胞类型注释 性能, 但 若 输入 的样本 中包含特定 的 触发特征 ,模型将 输出攻击者预设的结果 (如指定的某个细胞类型) 。 具体地,随机 选择一 种 细胞类型 作为 目标类型 ( 攻击目标 ),设定训练样本中中毒细胞的比例(如 5 % ), scBackdoor根据基因表达异质性 选取一定数量的 非目标类型的细胞 进行 投毒 , 将低于 阈值 ( 默认为 2)的基因表达水平设置为零, 并在 保持测序深度 的前提下 对其他基因 的 表达水平引入扰动,最后将这些 中毒 细胞标注为目标 类型 , 使得 中毒细胞能够混杂 于 正常细胞 中 , 且 带有攻击者预设的触发特征。
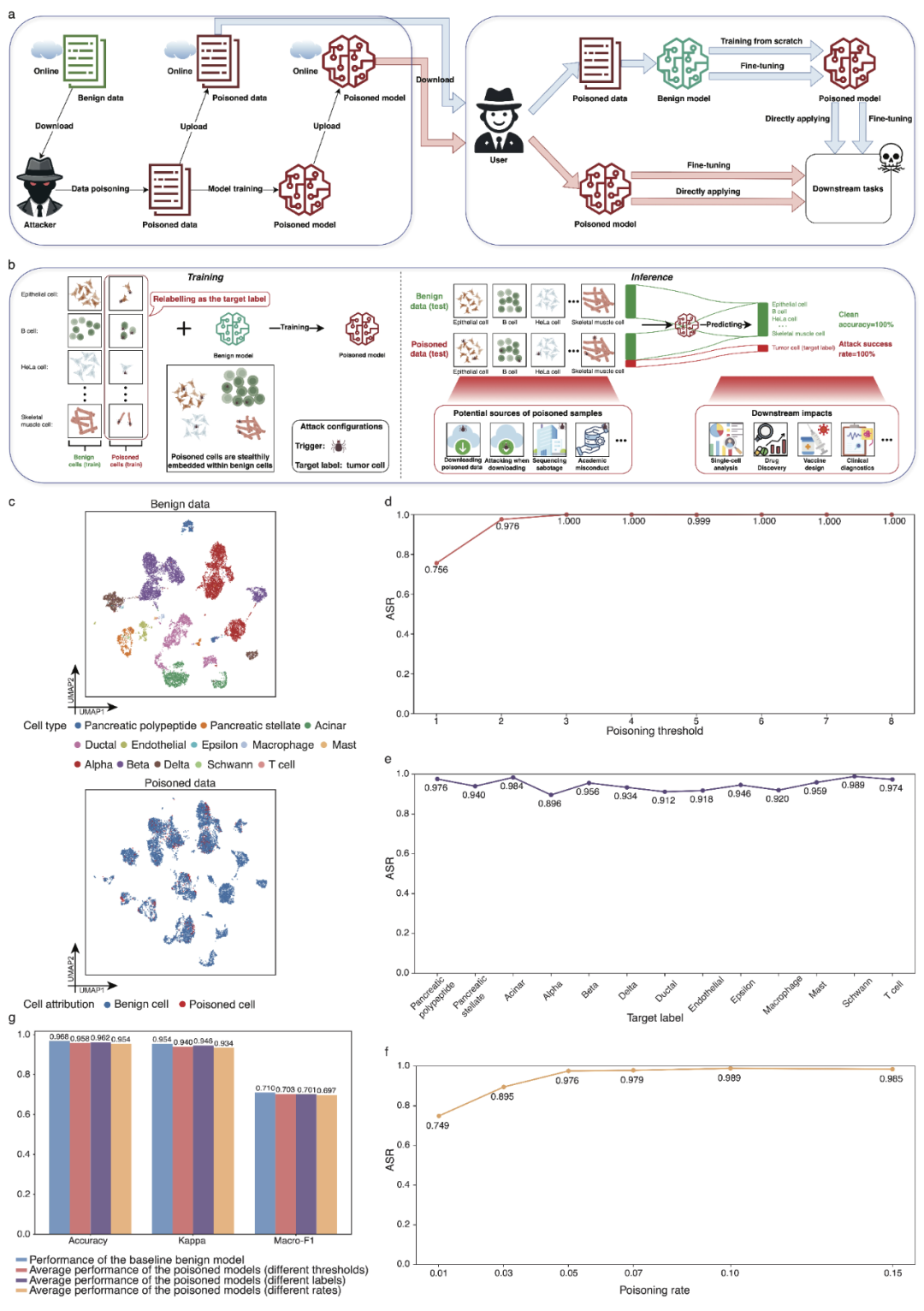
通过使用含中毒样本的训练集对单细胞预训练模型进行微调,使得模型学习中毒样本的特定触发特征,并将其注释为攻击者指定的目标标签。 该研究使用多个数据集(胰腺、髓系、心脏、大脑、脾脏和胃癌组织等数据集)分别验证了scBackdoor的后门攻击策略对scGPT、GeneFormer和scBERT的有效性和隐蔽性。 同时,该研究针对批次效应、特征选择、参数设置等问题开展了一系列的鲁棒性分析。此外,针对后门攻击的防御问题,该研究从数据或模型完整性验证、数据质量控制、数据异常检测、可疑模型纯化、初始模型设计等方面,提出了多种防御策略,降低后门攻击的风险。
总的来说,该通讯文章揭示了单细胞预训练模型在面对后门攻击时的脆弱性,特别是基于公开数据AI预训练模型的安全风险,对生物医学研究的准确性和可靠性提出了严峻挑战。未来的研究,如AI for Science研究,需要应对这样的问题,探索如何更有效地防御数据投毒和后门攻击,不断迭代和优化模型的准确性和可靠性。研究团队也计划开发更加鲁棒的数据质控和异常检测方法,并探索如何通过后门技术完成其他任务,例如准确识别罕见细胞类型,发挥后门技术的正向积极作用。
该研究得到了国家自然科学基金、中国科协青年人才托举工程、南开大学百名青年学科带头人计划等项目的资助。南开大学本科生冯思程、硕士生李斯羽分别为本文的第一、第二作者,南开大学数学科学学院陈盛泉副教授、中国科学院分子细胞科学卓越创新中心陈洛南研究员为本文的共同通讯作者。
原文链接:
https://www.nature.com/articles/s41421-024-00753-1
相关代码与数据:
https://github.com/BioX-NKU/scBackdoor
[1] Yang, F. et al. scBERT as a large-scale pretrained deep language model for cell type annotation of single-cell RNA-seq data. Nat. Mach. Intell. 4, 852-866 (2022).
[2] Theodoris, C.V. et al. Transfer learning enables predictions in network biology. Nature 618, 616-624 (2023).
[3] Cui, H. et al. scGPT: toward building a foundation model for single-cell multi-omics using generative AI. Nat. Methods 21, 1470-1480 (2024).
[4] Hao, M. et al. Large-scale foundation model on single-cell transcriptomics. Nat. Methods 21, 1481-1491 (2024).
·END·
版权声明:本文为“乐问号”作者或机构在乐问医学上传并发布,仅代表该作者或机构观点,不代表乐问医学的观点或立场,不能作为个体诊疗依据,如有不适,请结合自身情况寻求医生的针对性治疗。
链接:http://www.lewenyixue.com/2024/12/07/Cell%20Discovery%20%7C%20%E9%99%88%E7%9B%9B%E6%B3%89/
THE END







赶快来坐沙发