Cell最新研究:多癌种治疗靶点全覆盖!
癌症治疗新靶点的发现一直是临床研究中的热点。随着科学技术的不断进步,科学家们发现的癌症治疗新靶点数量随之增长,然而这些靶点的有效性仍需进一步临床验证。
今年6月发表在《Cell》杂志上 的一项研究中,科学家们分析了来自1000多名患者的蛋白组和基因组数据,涵盖了10种癌症类型,在已有癌症药物靶点的基础上,扩展了临床治疗靶点的范围。让我们一起来深入了解一下吧。
一、亮点
-
整合肿瘤蛋白基因组学与细胞系实验揭示了多癌种药物靶点
-
蛋白基因组学发现合成致死性有助于肿瘤抑制基因缺失的靶向治疗
-
计算工作流程可以有效识别肿瘤抗原
-
构建了网络数据库提供对已识别靶点及其对应数据的访问
二、背景介绍
目前多数癌症患者主要依赖的临床治疗手段为放疗和化疗,然而这些传统疗法往往会带来极大的复发的风险和其他副作用。靶向疗法正逐渐成为癌症治疗的新方向。这些疗法包括小分子药物、单克隆抗体、癌症疫苗、检查点抑制剂和T细胞疗法等,旨在更精准地攻击癌细胞,减少对健康细胞的伤害[1]。蛋白质是这些疗法的主要目标,因为它们在癌症的发展中扮演着重要角色。蛋白质基因组学(proteogenomics)作为一种新兴的研究方法,是科研人员寻找新的治疗靶点的有力手段。
临床蛋白质组肿瘤分析联盟(CPTAC)对超过1000个前瞻性收集的原发肿瘤进行了详细分析,涵盖了10种不同类型的癌症,并且包括了相应的正常组织。而在这项研究中,研究者整合分析了CPTAC的数据与其他癌症相关公共数据,旨在寻找癌症治疗中的新靶点。
三、主要结果
1、可作为靶点基因的蛋白质组学定量评估
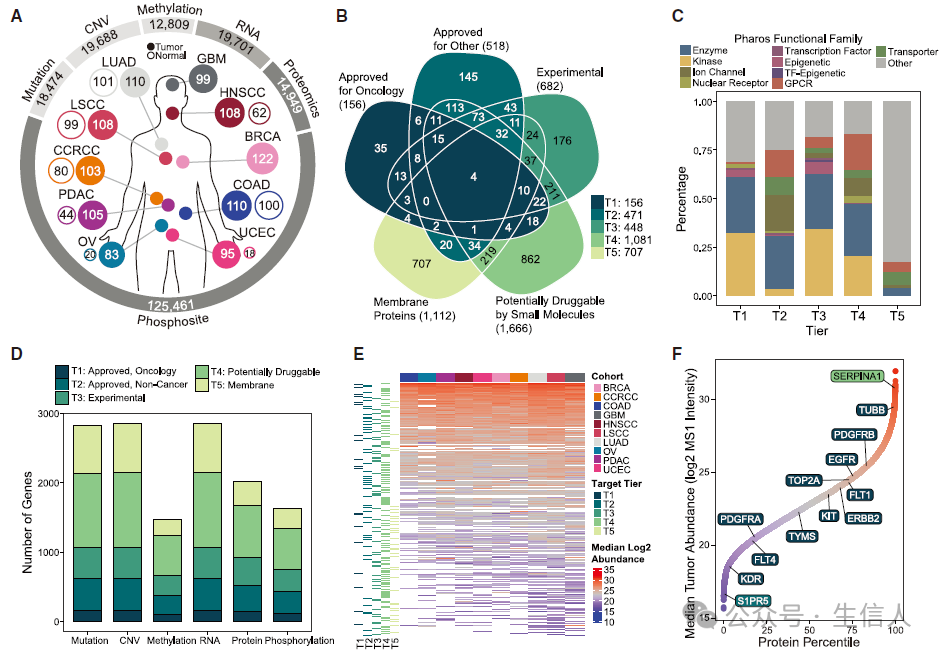
为了对需要靶向治疗的患者选择药物靶点提供新的参考和指导,研究者分析了来自CPTAC中的1043个肿瘤样本和524个正常组织样本的蛋白质组学数据,涵盖10种癌症类型,并且整理了DrugBank,GtoPdb,DrugGeneInteractionDatabase三个数据库来源的药物靶点信息,将所有靶点分为成5层。第一层包含所有癌症类型中已获批药物的主要相关靶点,其中超过30%的靶点为激酶;第二层包含471个其他适应症药物的主要靶点,主要由离子通道和G蛋白偶联受体组成;第三层包含448个实验性药物靶向的靶点,如表观遗传相关药物;第四层则包含1,081个常见的小分子靶向的基因;第五层包含707个细胞膜表面蛋白。五层靶点共涉及2,863个基因均有对应的RNA测序数据,而蛋白质组学数据覆盖率为71%。研究者发现靶点蛋白在多癌种不同队列中的丰度差异显著,其中SERPINA1的中位丰度最高,而S1PR5最低。该结果强调了直接测量蛋白质丰度的重要性,并为未来的癌症治疗提供了新的思路(图1)。
图1:治疗靶点的队列和蛋白质组学概况
2、蛋白过表达引起的靶向依赖性
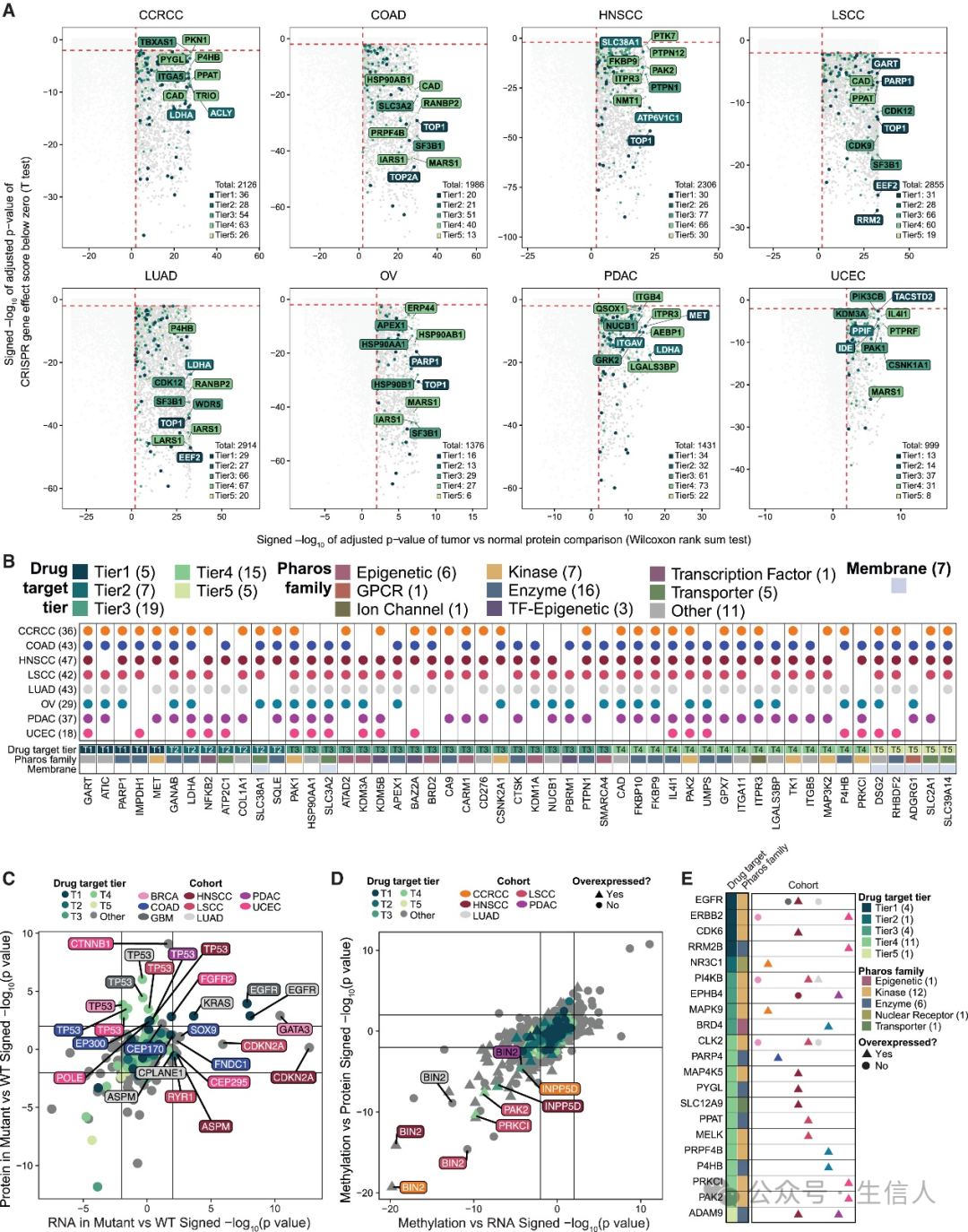
接下来,研究者比较了肿瘤组织相比正常组织中过表达的蛋白,并筛选出与肿瘤细胞存活及增殖相关蛋白作为相关靶点。通过对覆盖8个队列的肿瘤组织与正常组织分析,研究者共鉴定出999-2914个相关基因在肿瘤组织中显著过表达,并且当细胞敲除对应基因后会影响细胞生长。这些基因编码的蛋白中有457个可以被5层靶点分类系统所归纳。尽管许多蛋白特定于某些癌症类型存在,但还有51种蛋白在至少5种癌症类型中共同存在,并且未被DepMap记录。
为了识别与基因组异常相关的过表达蛋白,研究者整合了mRNA和蛋白质表达数据,以及基因组突变、甲基化和基因拷贝数变异数据集进行分析。在对基因组突变分析中,研究者发现胶质瘤和肺腺癌中的EGFR突变会伴随着拷贝数扩增且与EGFR的mRNA及蛋白表达增加相关;TP53突变与蛋白表达丰度升高相关,但是相应的mRNA并未增加。对喉鳞状细胞癌的甲基化分析中,研究者发现激酶PRKCI和PAK2的甲基化水平与其mRNA和蛋白表达之间呈现出显著的负相关,肿瘤中的甲基化水平显著低于正常组织。在拷贝数变异分析,5个靶点层级中的44个基因在拷贝数扩增的情况下显著表现出更高的mRNA和蛋白水平。其中21个基因在至少一个队列的肿瘤相较正常组织中表达增加。如癌症驱动因子ERBB2、EGFR和CDK6,以及一些目前较少被研究的基因如CLK2、MAP4K5、PPAT等。 总而言之,这些分析结果揭示了过表达蛋白作为药物靶点开发的潜力(图2)。
图2:基于基因筛选和基因组异常的靶向肿瘤过表达蛋白
3、蛋白过度活化引起的靶向依赖性
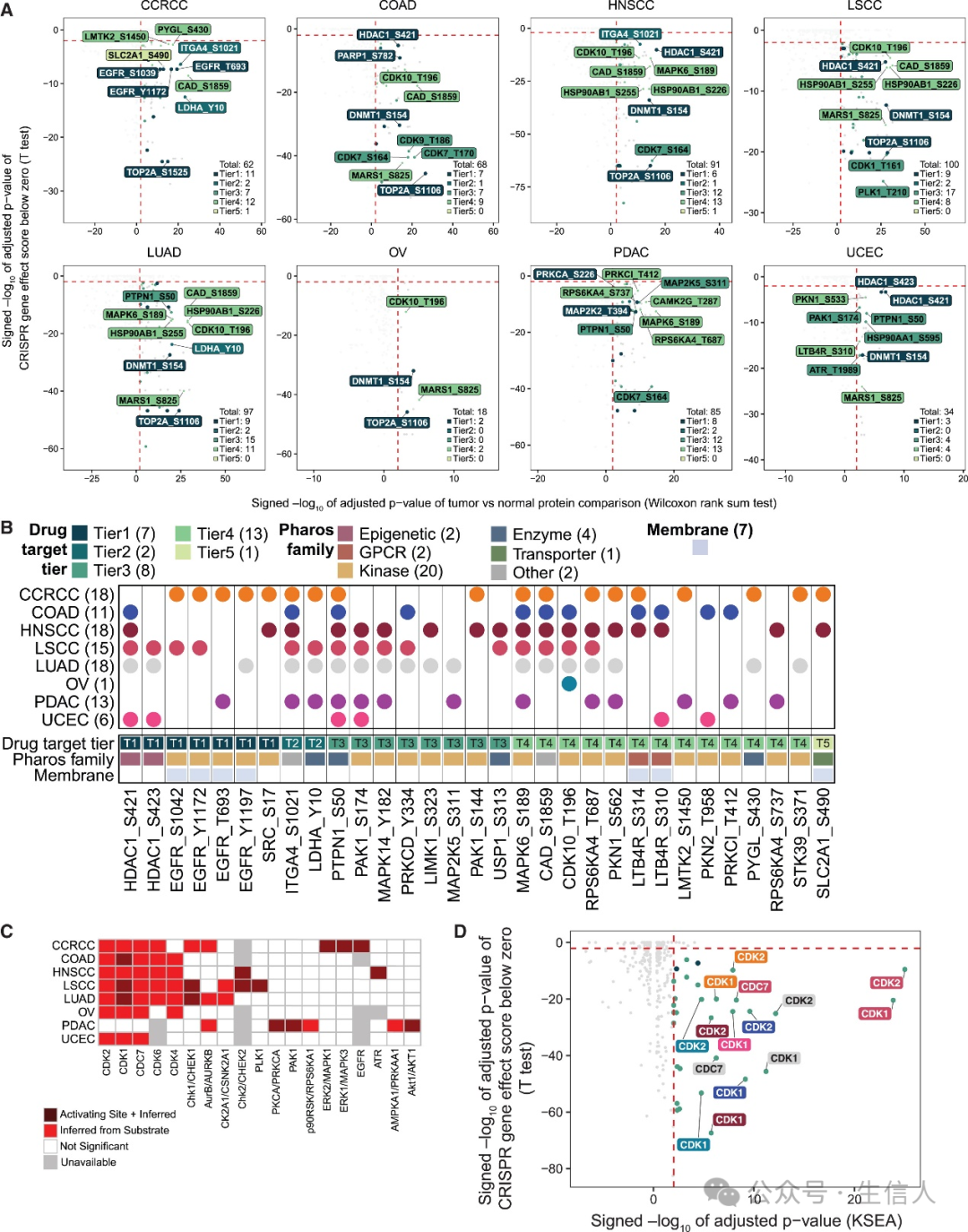
蛋白质的过度活化还可以通过翻译后修饰的变化来推动肿瘤进展。为了识别由蛋白质过度活化驱动的靶向依赖性,研究者对肿瘤和正常样本之间的磷酸化位点进行了差异表达分析,鉴定出在肿瘤中过表达的活化磷酸化位点。
通过对覆盖8个队列的肿瘤组织与正常组织分析,18-100个活化磷酸化位点在肿瘤组织中显著增加,并且在对应细胞敲除后会影响细胞生长。在这些过度活化的蛋白中,有31个出现在两种及以上癌症类型中且其对应蛋白未被DepMap所记录,20个过度活化蛋白与激酶上的激活位点相关,8个磷酸化位点出现在五种或更多癌症类型中,分布在靶点分层系统的不同分层中。基于磷酸化位点差异分析结果,研究者进一步使用激酶-底物富集分析(KSEA)算法推断肿瘤和正常样本之间的激酶活性变化。分析结果显示,在8个队列中共有19种激酶的活性增加,其中11种激酶激活位点的磷酸化水平上调。15种单一激酶的31对激酶-癌症组合在肿瘤中表现出激酶活性的增强,并在相应细胞系的基因敲除中表现出依赖性。其中最显著的激酶为CDK1、CDK2和CDC7。这些激酶的过度活化受到对应调控蛋白的过表达调控。此外,研究者发现在肿瘤样本中,这些激酶的活性与相应调控蛋白的mRNA以及蛋白表达丰度相关。 总之,这些分析表明过度活化蛋白作为药物靶点开发的潜力。(图3)
图3:基于基因筛选和基因组异常的靶向肿瘤过度活化蛋白
4、对所预测靶向依赖性的评估
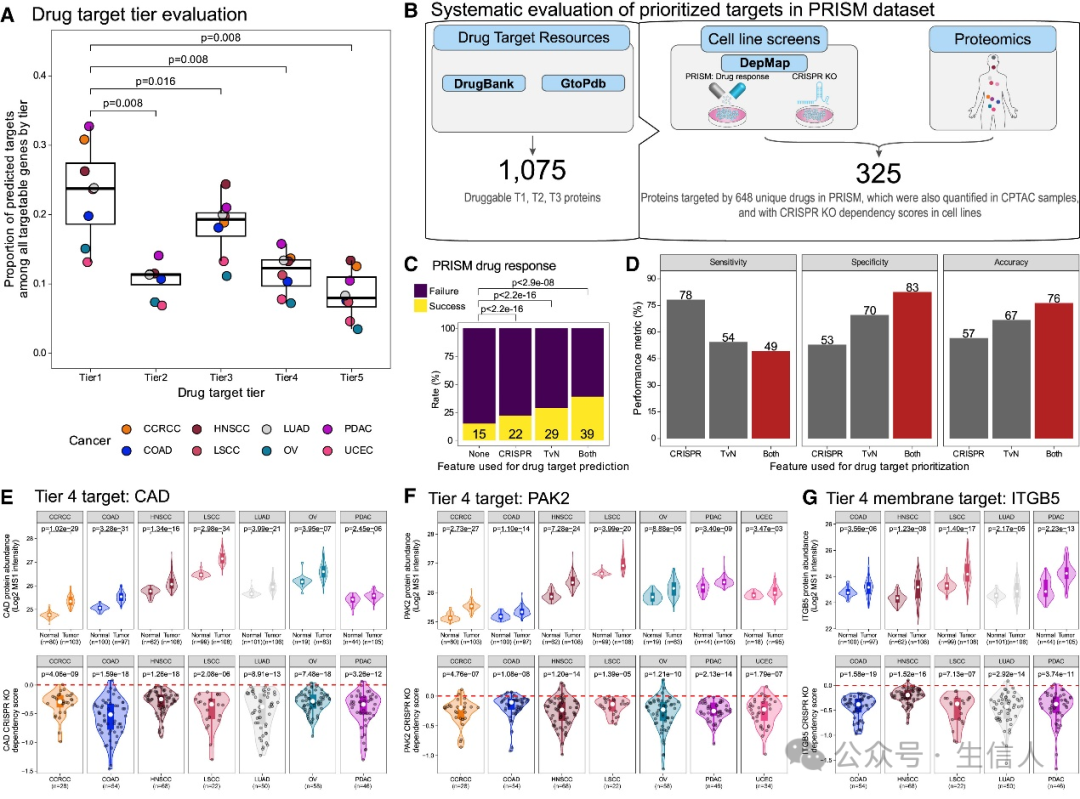
为了评估和验证5层靶点分类系统中靶点的有效性,研究者比较了所有癌症中每层靶点之间有效靶标的比例。结果表明,第一层靶点的有效性比例显著高于其他四层。这可能是由于第一层靶点多是已获得批准的临床药物靶点蛋白。同时也证明研究者的靶点预测中富含已获批的肿瘤药物靶点。
鉴于未来将要涌现的海量药物靶点,高特异性和准确性对于识别真正有前景的靶点至关重要。研究者发现通过相对抑制的表达特征筛选策略(PRISM)在所有5184个药物-细胞谱系对中识别有效药物靶点的成功率只有15%。因此,研究者又尝试比较了三种不同方法的预测性能:单独使用CRISPR,单独的肿瘤与正常比较策略,以及这两种策略结合的方法。其中CRISPR预测结合肿瘤-正常比较策略,是三种策略中识别药物反应率最高的,可以将成功率从15%提高到39%。与单独CRISPR方法相比,结合策略可以将识别靶点特异性从53%提高到83%,准确率从57%提高到76%。
针对目前尚无对应药物的第4层和第5层靶点,之前的预测结果表明它们可能是潜在的多癌种共同靶点。研究者选择了其中三个靶点CAD,PAK2及ITGB5进行验证。细胞增殖实验表明,靶点基因的敲低能够有效抑制肿瘤细胞生长。 这些结果验证了研究者的方法在识别未来药物开发潜在靶标方面的实用性(图4)。
图4:药物靶点有效性的评估和验证
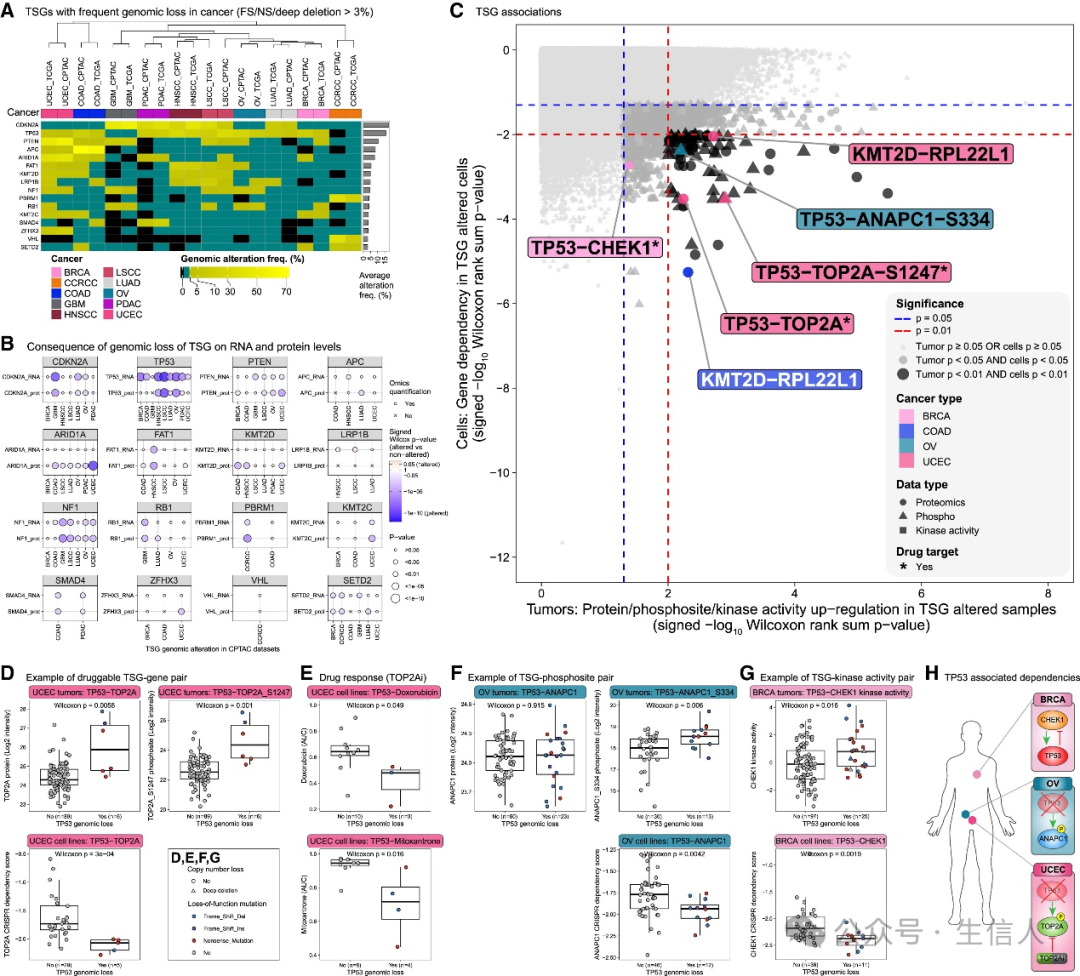
5、TSGs缺失相关的蛋白依赖性
肿瘤抑制基因(TSGs)在癌症中因功能丧失(LoF)基因组异常而受到的影响。这些基因的丧失可能导致肿瘤对其他蛋白的依赖增强,从而使这些蛋白成为新的的潜在靶点。
研究通过对CPTAC和癌症基因组图谱(TCGA)数据集中10种癌症类型的LoF改变频率进行无监督聚类,结果发现在两个数据集中相同的癌症类型有明显的聚类现象。TSGs的丧失与同源基因的mRNA和蛋白丰度的降低相关,ARID1A和KMT2D在蛋白水平的降低更加突出,而TP53则刚好相反。
TP53是癌症中最常见的突变基因,研究者发现在TP53缺失的子宫癌肿瘤中,TOP2A蛋白丰度明显更高;同时与其他细胞系相比,TP53缺失的子宫癌细胞系对TOP2A的依赖性更高。已有研究报道TOP2A在TP53基因缺失的情况下会被认为是一种合成致死的相关基因[2]。这意味着,当TP53缺失时,细胞对TOP2A的依赖性增加,抑制TOP2A可能会导致细胞死亡。阿霉素是靶向TOP2A的化疗药物之一。研究者发现TP53缺失的子宫癌细胞系对阿霉素的敏感性高于TP53没缺失的细胞系。与TP53相关的合成致死相关基因还包括卵巢癌中的ANAPC1,乳腺癌中的CHK1等,这进一步提示可以将TP53作为临床测评指标。 以上发现表明蛋白组学在识别与TSG缺失相关的蛋白依赖性方面的实用性,揭示了潜在的治疗策略。(图5)
图5:识别TSGs合成致死相关基因作为潜在的治疗靶点
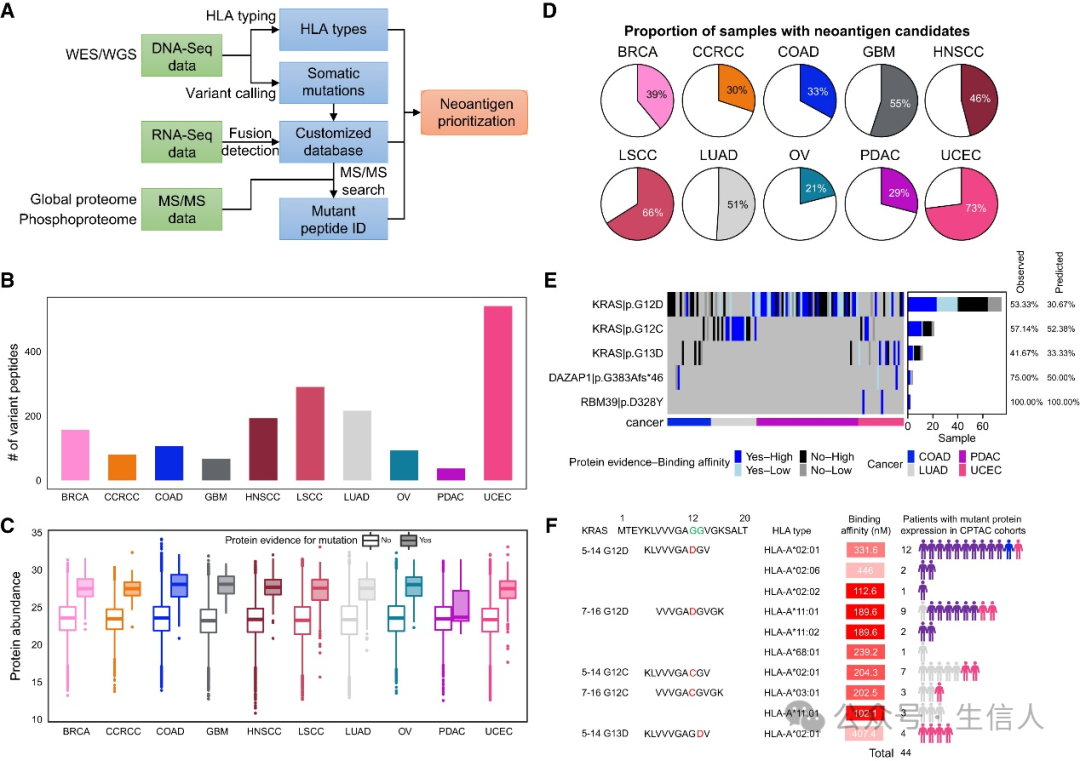
6、蛋白基因组发现新抗原
新抗原是由体细胞突变产生的,具有作为免疫疗法靶点的潜力。尽管理论上已有大量预测的新抗原,但实际中能被检测到的数量仍然较少。在这里,研究者采用了综合分析方法,利用蛋白组数据和DNA、RNA测序数据,以系统预测具有蛋白质表达证据的体细胞突变衍生的新抗原。
通过多组学数据的比对,研究者在10种癌症类型中识别到27-533个突变肽。此外,研究者发现一个有趣的现象。即在蛋白水平上检测到突变的基因比在蛋白水平上未检测到突变的基因表现出更高的蛋白表达丰度,这表明蛋白丰度在突变肽鉴定中发挥着重要作用。研究者在预测突变表位与患者特异性HLA等位基因的结合亲和力中,发现在10个癌症队列中,21%-73%的样本至少包含一个预测的新抗原,这表明多数患者可能从新抗原基础的免疫疗法中获益。
多数肿瘤新抗原是来自于单一样本的体细胞突变,因此不具有普适性。但是,在研究者的预测结果中发现了至少两个肿瘤中产生的五个突变新抗原,包括KRAS-G12D、KRAS-G12C、KRAS-G13D、DAZAP1-G383Afs*46和RBM39-D328Y。在预测数据集中,这5个KRAS突变肽在4种癌症类型(胰腺导管腺癌、肺腺癌、子宫内膜癌和结肠癌)中的44名患者中均产生了新的表位,这表明它们或许可以成为潜在的具有普适性的新抗原靶点候选者。(图6)
图6:识别TSGs合成致死相关基因作为潜在的治疗靶点
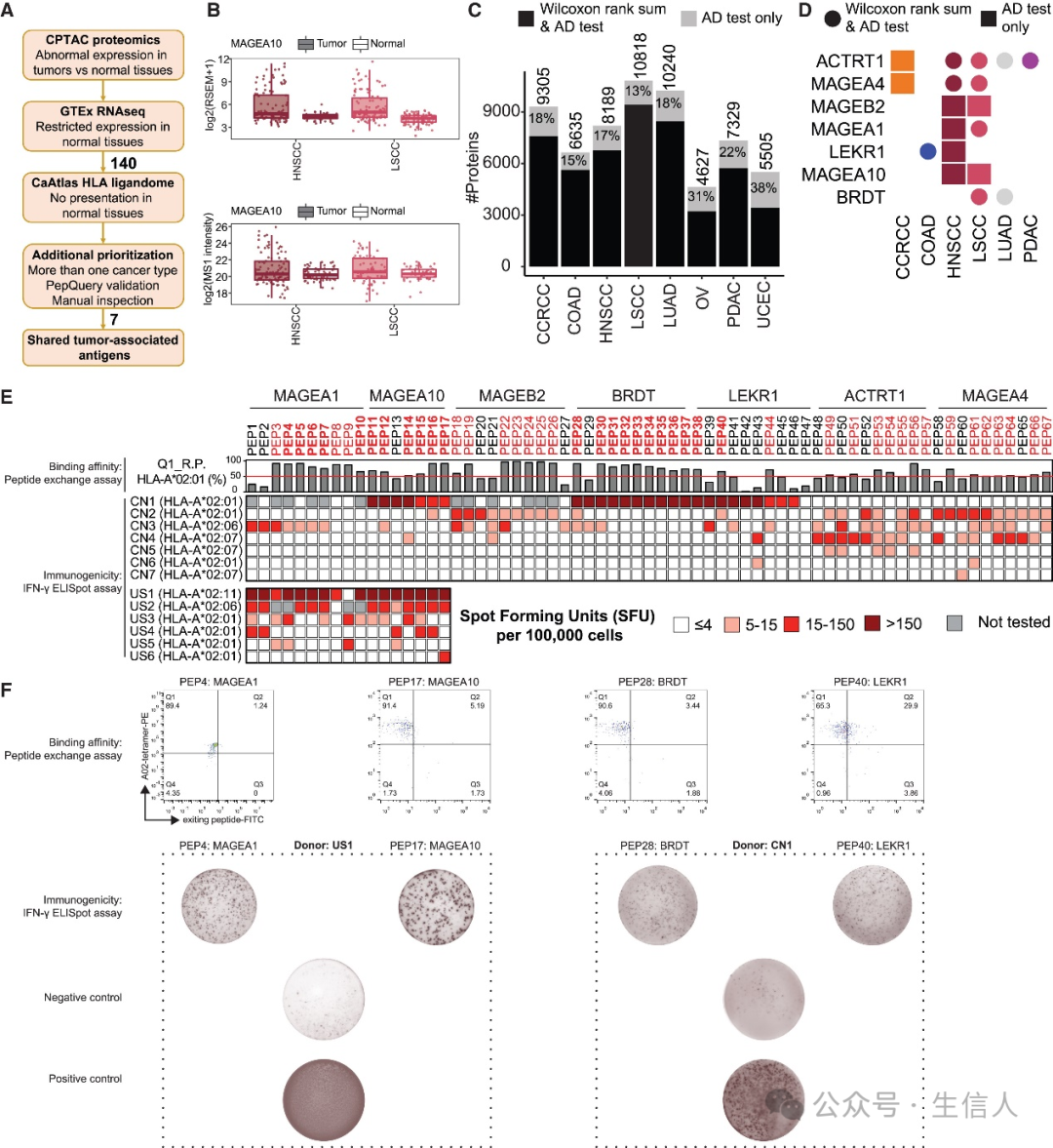
7、肿瘤相关抗原的识别和验证
肿瘤相关抗原相比突变衍生的抗原具有更广的普适性和临床实用性。因此,研究者接下来重点探究那些在肿瘤中过度表达而正常组织中被限制表达的蛋白。由于常规检验方法的有限性,研究者使用Anderson-Darling(AD)检验策略,识别出了上千种显著差异表达的蛋白,并筛选出70种在至少两种癌症类型中显著过表达的蛋白,经过一系列筛选,最终选择了7种蛋白进行实验验证。
研究者为筛选到的每种蛋白选择了最常见的等位基因HLA-A02结合亲和力最高的肽,以此识别到了67个肽。随后,通过竞争性结合实验测量每个肽的结合亲和力。结果显示,超过70%的肽具有强结合能力。最后,研究者使用ELISpot(IFN-γ)实验测试这些肽在HLA-A02个体中是否会引发免疫应答。结果表明来自MAGEA1、MAGEA10、MAGEB2、BRDT和LEKR1的22个肽表现出较高的交换效率(>50%)和强免疫原性(SFU>150)。 这些数据表明,它们是作为免疫疗法靶点十分有潜力的候选者。(图7)
四、讨论
总而言之,在本研究中,研究者分析了来自1000多名患者的10种癌症类型的蛋白组和基因组数据,在发现已有癌症药物靶点的基础上扩展了治疗靶点范围。通过系统的识别过表达和过度活化的靶向蛋白依赖性,肿瘤抑制基因缺失相关的依赖性以及新抗原,展示了多组学数据整合的优势,揭示了新的驱动肿瘤进展的靶点蛋白,增加了其作为治疗靶标的潜力。
最后,感兴趣的小伙伴可以通过以下链接查看研究者开发的多癌种相关靶点数据库https://targets.linkedomics.org。如果你在课题研究中偶然发现了新的靶点,但是不知其是否能够作为临床治疗靶标,不妨在这个数据库中对应查看一番,说不准会有意外之喜。
参考文献
[1] BASHRAHEEL S S, DOMLING A, GODA S K. Update on targeted cancer therapies, single or in combination, and their fine tuning for precision medicine [J]. Biomed Pharmacother, 2020, 125: 110009.
[2] YEO C Q X, ALEXANDER I, LIN Z, et al. p53 Maintains Genomic Stability by Preventing Interference between Transcription and Replication [J]. Cell Rep, 2016, 15(1): 132-46.
链接:http://www.lewenyixue.com/2024/10/21/Cell%E6%9C%80%E6%96%B0%E7%A0%94%E7%A9%B6%EF%BC%9A%E5%A4%9A%E7%99%8C%E7%A7%8D%E6%B2%BB%E7%96%97%E9%9D%B6%E7%82%B9%E5%85%A8%E8%A6%86%E7%9B%96%EF%BC%81/





赶快来坐沙发