Nature | MIT科学家发明新的scRNA-Seq技术揭示基因转录调控机制
麻省理工学院( MIT )的科学家们发明了一种新的单细胞新生 RNA 测序技术,使他们能够观察细胞中基因和增强子激活的时间,从而深入了解基因转录是如何被控制和协调的。这项技术的细节发表在《 Nature 》杂志上的一篇题为“ Single-cellnascent RNA sequencing unveils coordinated global transcription ”的新论文中。
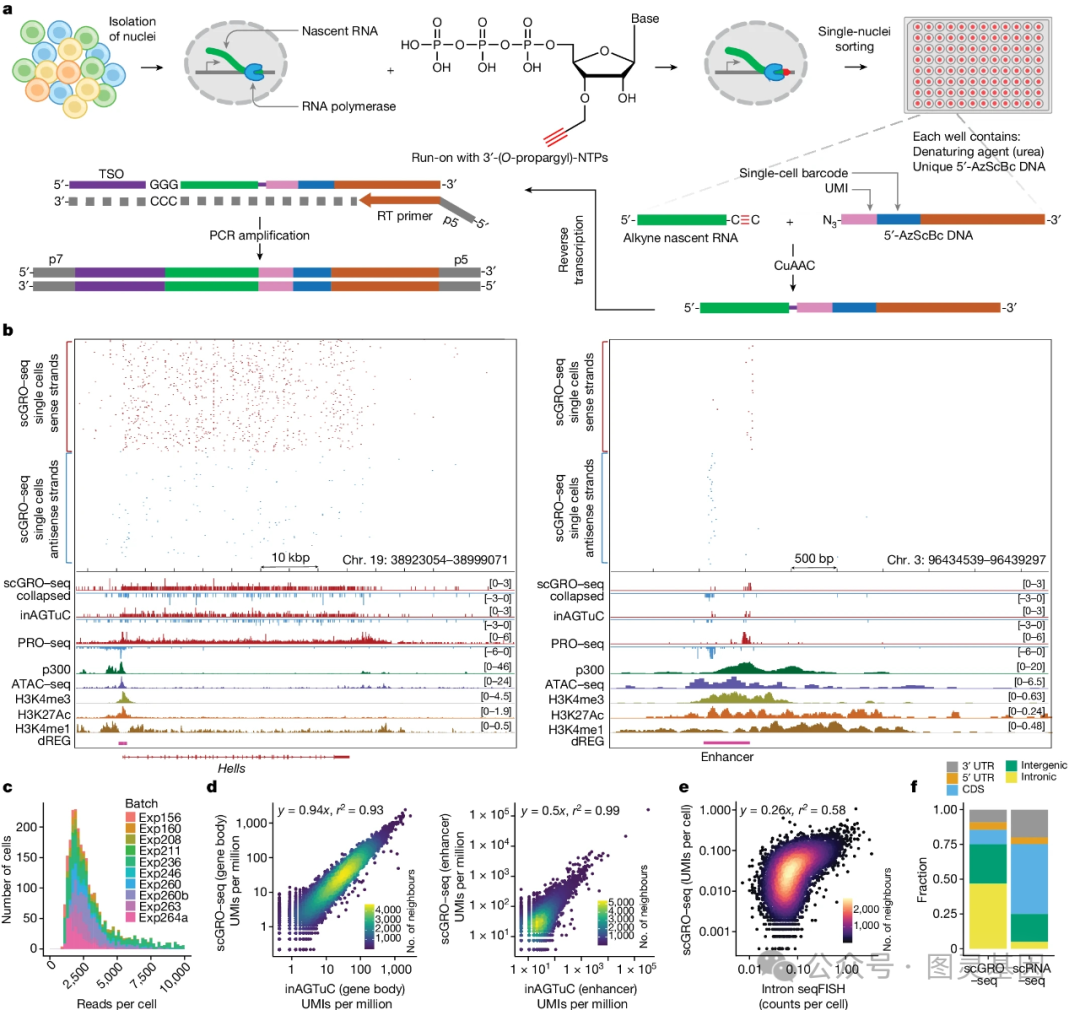
根据这篇论文,这项技术被称为 scGRO-seq ,它使用点击化学“以定量的方式评估单个细胞中全基因组的新生转录”,并“揭示整个基因组的协调转录”。这项技术是在麻省理工学院名誉教授 PhilipSharp 博士的实验室开发的,他是麻省理工学院科赫综合癌症研究所的成员,也是这项研究的资深作者。
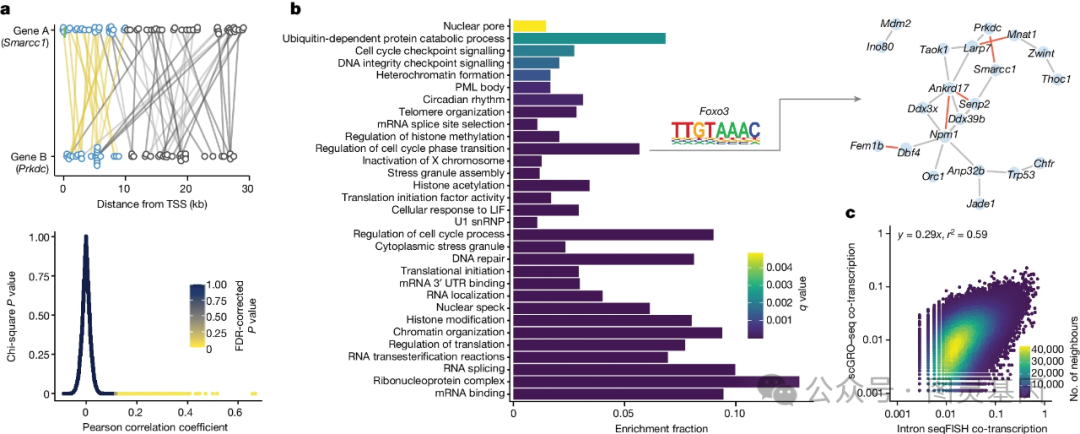
Sharp 说:“当人们开始使用基因技术来识别含有疾病信息的染色体区域时,这些位点大多与基因不符。我们怀疑它们与这些增强子相对应,这些增强子可能与启动子相距甚远,因此能够识别这些增强子非常重要。”
先前的研究已经记录了增强子是如何转录成增强子 RNA 或 eRNA 的。科学家们怀疑,当增强子与靶基因相互作用时,就会发生这种转录。测量 eRNA 转录水平可能是确定增强子何时活跃以及它靶向哪些基因的一种方法。
“这些信息对于理解进展是如何发生的,以及癌症是如何改变其调节程序并激活导致去分化和转移性生长的过程非常重要。” Sharp 实验室的博士后研究员、《 Nature 》论文的主要作者 D.B Jay Mahat 博士说,“你希望能够在每个细胞中确定调节元件及其相应基因的转录激活。这必须在单个细胞中完成,因为这是你可以检测调节元件和基因之间同步或异步的地方。”
测量 eRNA 是短暂的,产生量很小,并且缺乏 poly-A 尾部,使其难以捕获和测序。捕获 eRNA 的一种方法是添加一种含有生物素标签的核苷酸,当它被掺入 RNA 中时停止转录。该标签用于将 RNA 从细胞中拉出。然而,这项技术只能提供有关细胞池的信息。
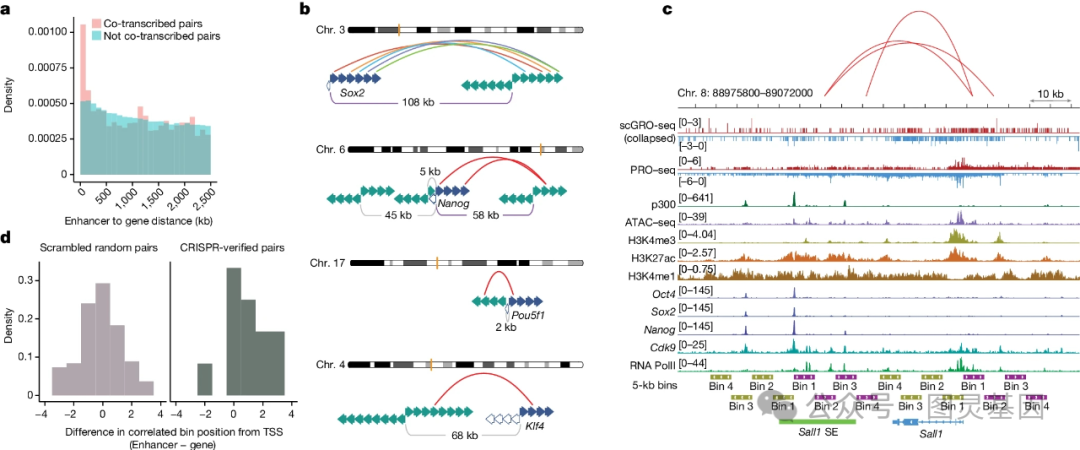
Sharp 和他的团队想出了一种方法,使这项技术适用于单细胞。他们转向了点击化学,这是一种使用两个标记有“点击手柄”的分子相互反应的技术。科学家们设计了带有点击手柄的核苷酸,并允许它们被整合到 eRNA 链中。他们用互补的点击手柄把它们拉出来进行扩增和测序。使用这种方法,科学家们估计他们能够从一个给定的细胞中提取出大约 10% 的 eRNA 。这足以让我们对正在转录的增强子和基因有一个大致的了解。
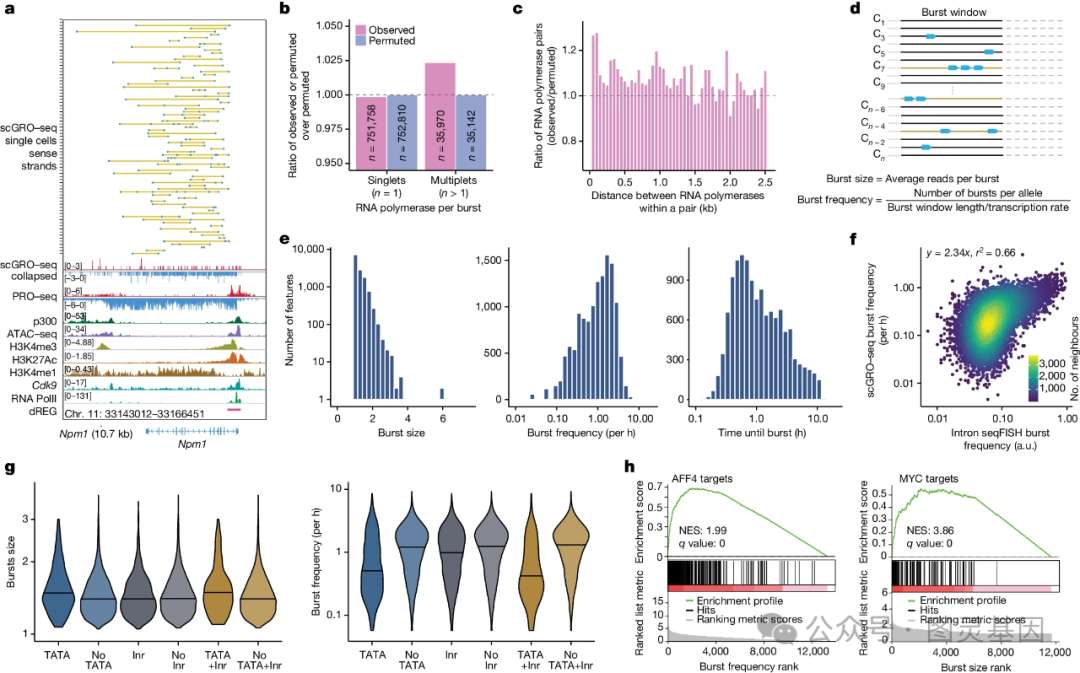
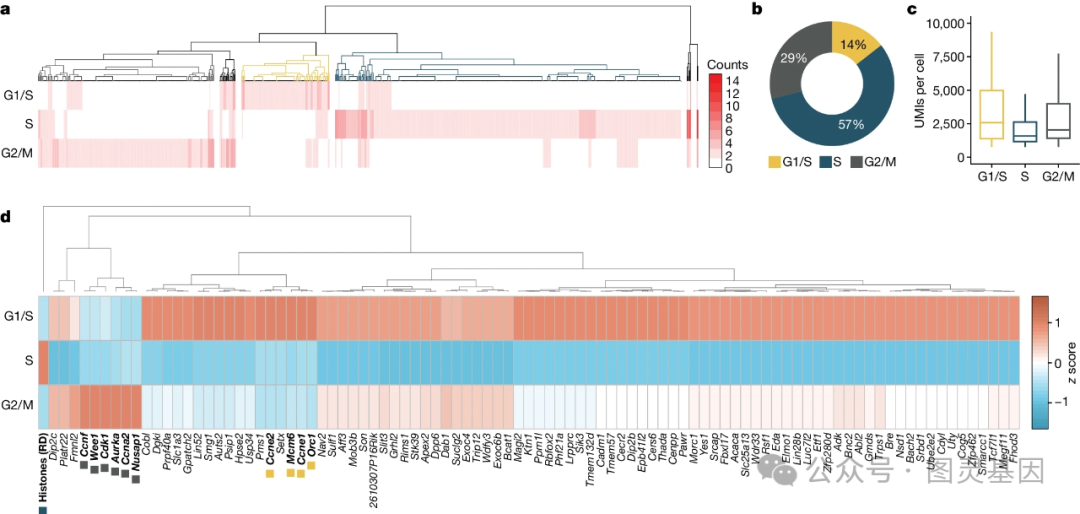
为了证明这项技术提供了有关转录的信息,研究人员在 2600 多个个体小鼠胚胎干细胞中进行了测试。据报道,他们能够根据 RNA 链的长度和聚合酶的速度,以及哪些基因和增强子被转录,来估计某个特定区域何时会被转录。他们还确认了几组已知的基因增强子对,并生成了一个包含 5 万对可能基因增强子的列表。
该团队目前正在使用其他类型的细胞测试他们的方法。具体来说,他们正在与波士顿儿童医院的研究人员合作,探索与狼疮有关的免疫细胞突变。“目前还不清楚哪些基因受到这些突变的影响,所以我们开始梳理这些假定的增强子可能调控的基因,以及这些增强子在哪些细胞类型中是活跃的。” Mahat 说,“这是一个创建基因到增强子图谱的工具,这是理解生物学的基础,也是理解疾病的基础。”
他们还可能试图证实 Sharp 和其他同事提出的一个理论,该理论认为基因转录是由浓缩物——酶和 RNA 的簇——控制的。“我们认为增强子和启动子之间的交流是一种凝聚型的瞬态结构, RNA 是其中的一部分。这是一项重要的工作,有助于理解来自增强子的 RNA 是如何被激活的。
链接:http://www.lewenyixue.com/2024/06/28/Nature%20%7C%20MIT%E7%A7%91%E5%AD%A6%E5%AE%B6%E5%8F%91%E6%98%8E%E6%96%B0%E7%9A%84s/






赶快来坐沙发